Original Article
Reference gene selection for gene expression study in tissues of long-tailed chickens
Yunjeong Noh1
,
Woncheoul Park1
,
Han-Ha Chai1,2,*
,
Dajeong Lim1,*
Author Information & Copyright ▼
1National Institute of Animal Science, Rural Development Administration, Wanju 55365, Korea
2College of Pharmacy, Chonnam National University, Gwangju 61186, Korea
*Corresponding author: Han-Ha Chai, National Institute of Animal Science, Rural Development Administration, Wanju 55365, Korea, Tel: +82-63-238-7306, E-mail:
hanha@korea.kr
*Corresponding author: Dajeong Lim, National Institute of Animal Science, Rural Development Administration, Wanju 55365, Korea, Tel: +82-63-238-7307, E-mail:
lim.dj@korea.kr
© Research Institute of Veterinary Medicine, Chungbuk National University. All rights reserved.
Received: Sep 29, 2020; Accepted: Oct 27, 2020
Published Online: Dec 31, 2020
Abstract
Quantitative real-time polymerase chain reaction (RT-qPCR) is a rapid and precise method of analysis to quantify the level of gene expression and is widely used in the diagnosis of diseases and quantitative analysis of genes. In RT-qPCR analysis, a reference gene (or housekeeping gene) is used for normalization of experimental results. Since this method of analysis detects a small quantity of the product, it is highly sensitive and it is important for the accuracy and reproducibility of the experiment to select a reference gene suitable for gene expression studies. As the expression levels of the reference gene are affected under different conditions, in order to determine the suitability of the housekeeping gene used as the reference gene, it is necessary to verify the expression stability. In the current study, the stability of the expression of 11 housekeeping genes (B2M, SDHA, GAPDH, RPL13, VIM, EEF1A1, HPRT1, GUSB, RPL19, ACTB, and ABL1) was investigated in the tissues of long-tailed chickens (heart, thigh, and breast). Expression stability evaluation was analyzed with four software: BestKeeper, NormFinder, geNorm, and RefFinder. In our study, GAPDH in heart tissue, HPRT1 in thigh tissue, and RPL13 in breast tissue were selected as the most stable reference genes. Evaluation of the expression stability of housekeeping genes can provide important data in gene expression studies by selecting an appropriate reference gene according to various conditions.
Keywords: RT-qPCR; housekeeping gene; BestKeeper; geNorm; NormFinder
Introduction
정량적 실시간 중합효소연쇄반응(quantitative real-time polymerase chain reaction, RT-qPCR)은 실시간으로 증폭되는 산물의 양을 측정하여 목적 유전자(target gene)의 발현 수준을 정량화하는 방법으로, 효율적이고 정확도가 높아 유전자 발현 검증, 유전자 서열 변이 확인과 같은 SNP typing, 생체시료부터 바이러스 검출 등 다양한 유전체 연구에 많이 적용되고 있다[1]. RT-qPCR은 형광물질을 통해 증폭된 산물을 정량적으로 검출하기 때문에, 민감도가 높아 샘플의 유형, RNA의 양과 품질, PCR의 효율 및 피펫팅(pipetting) 오류 등 여러 실험적 요소에 의해 실험결과의 정확도가 달라질 수 있다. RT-qPCR은 일정하게 발현되는 하우스키핑 유전자(housekeeping gene)를 기준 유전자(reference gene)로 하여 동일시료로부터 같은 조건하에서 목적 유전자를 증폭시켜 상대적인 정량을 통해 목적 유전자의 발현 수준을 측정한다[2, 3]. 그러나 모든 조건에서 기준 유전자로 선택된 하우스키핑 유전자가 안정적인 발현을 나타내지 않으며, 실험 및 생물학적인 조건에 따라 발현이 달라질 수 있기 때문에, RT-qPCR 분석에서 실험 조건별로 적합한 기준 유전자의 선정이 중요하다[4, 5].
긴꼬리닭(Gallus gallus var. domesticus)은 우리나라 재래닭에 포함되며, 국제연합 농업식량기구(http://dad.fao.org/en/Home.htm, FAO)와 가축 다양성 정보시스템(http://dad.fao.org/dad-is/en/, DAD-IS)에 “Ginkkoridak”으로 등재되어 있으며, 다른 토종닭과 비교하여 수컷의 꽁지깃이 1 m 이상 길어지는 표현형 특징이 있다[6]. 닭은 척추동물의 생물 의학 모델 유기체로 분자 생물학과 질병 등을 분석하기 위한 생물 의학 모델로, 닭을 이용해 전염병, 암 연구, 유전자 조절 등 다양한 생물학적 연구를 수행하기 위해서는 유전자 발현 분석이 필수적이다[7]. 닭에 대한 RT-qPCR의 유전자 발현 연구에서 TBP(TATA box binding protein), GAPDH(glyceraldehyde-3-phosphate dehydrogenase), RPL32(ribosomal protein L32), B2M(β-2-microglobulin), UB(ubiquitin) [8–10] 등의 하우스키핑 유전자들이 조직 유형이나 실험 조건 등에 따라 발현 변화가 적어(높은 안정성) 목적 유전자의 발현 수준을 정규화(normaliztion)하는데 적합한 기준 유전자로 많이 적용된다. 그러나, 긴꼬리닭에서 발현 안정성이 높은 기준 유전자를 탐색하는 연구는 거의 알려지지 않았다. 특히, 긴꼬리닭의 조직 시료로부터 기준 유전자의 발현 안정성 평가는 RT-qPCR 분석에서 품종 특이적인 꽁지깃 성장 등과 관련된 목적 유전자의 발현 데이터를 정규화하는 데 기초 정보로 제공될 것이다.
본 연구는 긴꼬리닭의 3가지 조직(심장, 다리 근육, 가슴살)에서 B2M, SDHA(succinate dehydrogenase complex flavoprotein subunit A), GAPDH, RPL13(ribosomal protein L13), VIM(Vimentin), EEF1A1(eukaryotic translation elongation factor 1 α 1), HPRT1(hypoxanthine phosphoribosyltransferase 1), GUSB(glucuronidase-β), RPL19(ribosomal protein L19), ACTB(β-actin), ABL1(ABL proto-oncogene 1, non-receptor tyrosine kinase)을 포함하는 11개 하우스키핑 유전자 발현을 RT-qPCR을 통해 측정한 후, 발현 안정성을 분석하였다. 통계 분석은 엑셀 소프트웨어 BestKeeper, geNorm 및 NormFinder와 웹 기반 소프트웨어 RefFinder를 사용하였고, 4종류 알고리즘을 통해 각 조직에서 발현 안정성이 높은 하우스키핑 유전자의 순위를 정하였다.
Materials and Methods
RNA 추출법
실험에 사용된 재료는 저온 냉동고(–80℃)에 보관 중이던 긴꼬리닭 수컷 3수(개체 1, 2, 4번) 각 조직(심장, 다리 근육, 가슴살) 시료를 사용하였다. 보관된 조직을 막자사발에서 액체 질소로 급속냉각시킨 후 곱게 갈아서 Trizol(Ambion, USA) 1 mL에 넣고 200 μL chloroform (Sigma-Aldrich, St. Louis, MO, USA )과 함께 혼합(vortexing)하였다. 그리고 18,000 ×g에서 15분 동안 원심 분리해 투명한 상층액 부분을 새로운 튜브에 분주하였다. 상층액이 들어간 튜브에 동량의 isopropanol을 넣고 18,000 ×g에서 10분간 원심 분리 후 펠렛(pellet)을 제외한 상층액 부분을 제거하였다. 이 펠렛을 70% EtOH로 수세하고, 실온에서 10분간 건조하여 최종적으로 RNA를 얻었다. 추출한 RNA는 DEPC(diethylpyrocarbonate) water에 녹인 후 ND-1000 spectrophotometer(Nanodrop, USA)를 이용해 순도를 검사하였고, 2% Agrose gel(BioD, Korea)에서 전기영동하여 RNA의 degradation을 확인하였다.
하우스키핑 유전자의 프라이머 제작
본 연구에서는 닭에서 선행 연구된 11개 하우스키핑 유전자(B2M, SDHA, GAPDH, RPL13, VIM, EEF1A1, HPRT1, GUSB, RPL19, ACTB, ABL1)를 선별하였고, 서열 정보는 NCBI(National Center for Biotechnology Information)에서 각 유전자의 mRNA 서열을 수집하여 프라이머(primer)를 제작하였다. 기본 설정은 증폭 산물의 길이(100–170 bp), 프라이머 길이(20 mers), 어닐링 온도(annealing temperature) (60℃–61℃) 그리고 GC함량(55%–60%)으로 하여 Primer 3 Plus(http://primer3plus.com/cgi-bin/dev/primer3plus.cgi) 프로그램을 통해 제작하였다(Table 1). 각 프라이머의 PCR 효율값(Efficiency %)은 1, 101, 102, 103배씩 희석하여 표준 곡선을 통해 기울기(slope)값을 구했다(이때 Efficiency %는 (10(–1/slope) – 1) × 100값이다.)[11].
Table 1.
Primer list of housekeeping genes selected as reference genes
| Gene symbol |
Description |
RefSeq |
Sequence (5’→3’) |
Ampliconsize (bp) |
|
B2M
|
β-2-Microglobulin |
NM_001001750 |
F: CAAGGTGCAGGTGTACTCCC
R: CCAGTCGTCGTTGAAGGACA |
166 |
|
SDHA
|
Succinate dehydrogenase complex flavoprotein subunit A |
NM_001277398 |
F: ACCATTTACCACCCCAGCAG
R: AGGCAAAACGGGAATAGGCT |
107 |
|
GAPDH
|
Glyceraldehyde-3-phosphate dehydrogenase |
NM_204305 |
F: TCACAGCCACACAGAAGACG
R: TTTCCCCACAGCCTTAGCAG |
116 |
|
RPL13
|
Ribosomal protein L13 |
NM_204999 |
F: GGATCCCAGGCGAAGAAACA
R: TCCTTTCTTCGGTGCAGACG |
118 |
|
VIM
|
Vimentin |
NM_001048076 |
F: GCCAGATGCGTGAAATGGAG
R: TGGCGAGCCATTTCTTCCTT |
112 |
|
EEF1A1
|
Eukaryotic translation elongation factor 1 α 1 |
NM_001321516 |
F: GGTTACCCGGAAAGATGGCA
R: TTGAAGAGGCAGACGCAGAG |
109 |
|
HPRT1
|
Hypoxanthine phosphoribosyltransferase 1 |
NM_204848 |
F: CAGAGAGACTGGCACGTGAA
R: TGGGGATTGACTTGTCACTG |
147 |
|
GUSB
|
Glucuronidase-β |
NM_001039316 |
F: TGGGTGAATGGAGTGCAAGT
R: AACAGTGATGCGGCAGAGAA |
111 |
|
RPL19
|
Ribosomal protein L19 |
NM_001030929 |
F: GGAAGAGAAAGGGTACGGCC
R: GCGGTCGATCTTCTTGGACT |
116 |
|
ACTB
|
β-Actin |
NM_205518 |
F: TCTGTATGCCAACACAGTGCT
R: TCATTGTGCTAGGTGCCAGG |
101 |
|
ABL1
|
ABL proto-oncogene 1, non-receptor tyrosine kinase |
XM_015279734 |
F: GCCTTGTAGGGGAGAACCAC
R: ACTTGTTGTAGGCCAGGCTC |
141 |
Download Excel Table
cDNA 합성
cDNA 합성은 SuperScript® III First-Strand Synthesis System for RT-PCR (Invitrogen, Carlsbad, CA, USA) kit를 사용하였다. cDNA 합성단계에서 추출한 RNA이외 사용되는 시약들은 SuperScript® III First-Strand Synthesis System for RT-PCR (Invitrogen) kit에 포함된 시약이다. 추출한 RNA (40 ng) 4 μL, random hexamers (50 ng/μL) 1 μL, dNTP (10 mM) 1 μL, DEPC-treated water 4 μL로 제조한 반응용액 Ⅰ을 65℃에서 5분 동안 반응한 후, ice에서 1분 반응하였다. 그 후 10× RT buffer 2 μL, MgCl2(25 mM) 4 μL, DTT (0.1 M) 2 μL, RNaseOUT™ (40 U/μL) 1 μL, SuperScript® III Reverse Transcriptase (200 U/μL) 1 μL로 총 반응용액 10 μL를 만든 후 반응용액 Ⅰ과 함께 혼합하여 25℃에서 10분, 50℃에서 50분, 85℃에서 5분 반응 후 바로 ice에 반응하였다. 마지막으로 RNase H 1 μL 넣고 37℃에서 20분간 반응하여 RNA를 제거해 주고 –20℃에 보관하였다. 합성한 cDNA에서 각 프라이머의 유전자 증폭을 위한 PCR 반응은 GeneAmp PCR system 9700(Applied Biosystems, Foster City, CA, USA) 기기를 이용하였고, cDNA 2 μL, 10× Buffer 2 μL, dNTP (2.5 mM) 1.6 μL, forward/reverse 프라이머(10 pmol) 각 0.4 μL, HS Taq polymerase (2.5 units/μL) 0.6 μL와 멸균증류수 13 μL를 넣어 총 20 μL로 맞추었다. 반응조건은 95℃에서 11분 반응 후 94℃에서 30초, 60℃에서 30초, 72℃에서 45초를 37회 반복하였고, 60℃에서 30분 반응하여 8℃에서 보관하였다. PCR 산물은 2% Agarose gel에서 전기영동 과정을 통해 확인하였다.
Real-time quantitative PCR (RT-qPCR) 분석
유전자 발현 분석을 위한 RT-qPCR은 7500 Real-time PCR System(Applied Biosystems) 기기로 수행하였고, 반응용액은 cDNA 2 μL, 2X SYBR Green Master MIX (Applied Biosystemes) 10 μL, forward/reverse 프라이머(10 pmol) 각 1 μL 넣은 후 DEPC Water 6 μL를 마지막으로 넣어 총 20 μL를 만들었다. PCR조건은 50℃에서 2분, 95℃에서 10분간 반응 후 95℃에서 15초, 60℃에서 1분 동안 40회 반복하였다. 그리고 최종적으로 95℃에서 15초, 60℃에서 1분, 95℃에서 30초, 60℃에서 15초 반응시켰다. 각 프라이머의 유전자 발현 수준은 한계 사이클(Threshold cycle, Ct)을 통해 확인하고, 용융 곡선(Melting curve)을 통해 비특이적인 증폭 반응을 확인하였다.
하우스키핑 유전자의 발현 안정성 분석
유전자 발현 안정성 평가는 심장, 다리 근육, 가슴살로 구분하여 분석하였다. 분석 프로그램은 마이크로소프트-엑셀 기반 소프트웨어인 Bestkeeper [12], NormFinder [13], geNorm [14] 도구를 사용하였고, 마지막으로 세 가지 도구를 통합하여 모든 하우스키핑 유전자를 비교하는 웹-기반 소프트웨어 RefFinder(https://www.heartcure.com.au/reffinder/?type=reference#)를 통해 전체 안정성 순위를 정했다. Bestkeeper 분석은 각 유전자의 Ct값으로 표준 편차(standard deviation, SD)와 변동 계수(Coefficient of variation, CV)를 통해 발현 변이를 계산하며, 가장 낮은 표준편차값을 가지는 유전자를 최적의 기준 유전자로 나타내지만, SD > 1인 유전자는 일관성이 없는 것으로 간주한다[15]. NormFinder와 geNorm 분석은 bestkeeper와 달리 2−ΔCt값으로 유전자 안정성을 결정한다(이때 ΔCt = Ct값 – 최저Ct값[16] 공식을 이용해 상대 수량으로 Ct값을 변환함). geNorm 분석은 유전자 간의 비교를 통해 상호 안정성을 쌍별 변이(pairwise variations, V)로 결정하고, 각 유전자의 평균 발현 안정성(stability value, M)값을 통해 가장 낮은 M값을 높은 안정성으로 정한다. NormFinder는 그룹 내 및 그룹 간의 발현 변이를 기반으로 하우스키핑 유전자의 안정성값을 분석하여 최적의 기준 유전자 순위를 결정한다.
Results
PCR에서 하우스키핑 유전자들의 발현
각 유전자의 PCR 효율값은 81%–106% 사이로 나타났으며, 용융 온도(melting temperature)는 78℃–86℃ 사이로 나타났다(Table 2). 또한 전기영동에서 모든 하우스키핑 유전자가 단일 밴드의 형성을 보였으며(Fig. 1), RT-qPCR의 용융 곡선에서도 유전자의 비특이적인 증폭 반응은 보이지 않았다(Fig. 2, 3). 증폭된 유전자의 발현 수준을 나타내는 Ct값의 평균은 GAPDH가 19.27(심장), 16.47(다리 근육), 15.26(가슴살)으로 모든 조직에서 높은 발현 수준을 보였으며, 심장에서는 GUSB유전자가 평균27.73으로 발현 수준이 낮게 나타났고, 다리 근육과 가슴살에서 ABL1 유전자가 27.37(다리 근육), 27.07(가슴살)로 가장 낮은 발현 수준을 나타냈다.
Table 2.
Melt temperatures and efficiency values of housekeeping genes
| Gene |
Annealing temperature (°C) |
Melt temperature (mean ± SD, °C) |
Efficiency (%) |
|
B2M
|
60 |
85.5 ± 0.2 |
100 |
|
SDHA
|
60 |
79.1 ± 0.2 |
106 |
|
GAPDH
|
60 |
83.8 ± 0.2 |
91 |
|
RPL13
|
60 |
81.2 ± 0.2 |
94 |
|
VIM
|
60 |
79.4 ± 0.2 |
95 |
|
EEF1A1
|
60 |
81.9 ± 0.2 |
95 |
|
HPRT1
|
60 |
78.6 ± 0.2 |
104 |
|
GUSB
|
60 |
81.6 ± 0.2 |
101 |
|
RPL19
|
60 |
82.7 ± 0.2 |
100 |
|
ACTB
|
60 |
80.9 ± 0.1 |
94 |
|
ABL1
|
60 |
83.7 ± 0.1 |
81 |
Download Excel Table
Fig. 1.
Confirmation of specific amplification and size of each primer. Electrophoresis result on 2% agarose gel of amplified product. (A) heart, (B) thigh, (C) breast.
Download Original Figure
Fig. 2.
Melting curve of housekeeping genes amplified by RT-qPCR in long-tailed chicken thigh tissues. RT-qPCR, quantitative real-time polymerase chain reaction.
Download Original Figure
Fig. 3.
Melting curve of housekeeping genes amplified by RT-qPCR in long-tailed chicken tissues. (A) Heart (B) Breast. RT-qPCR, quantitative real-time polymerase chain reaction.
Download Original Figure
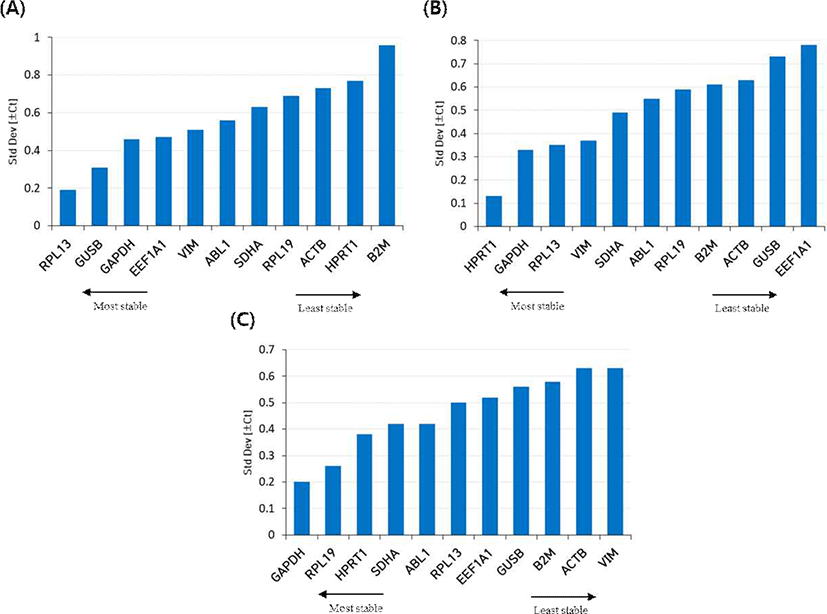
Bestkeeper 분석
이 소프트웨어 도구는 각 유전자의 평균 Ct값을 기반으로 SD와 CV 등의 값이 측정되며(Table 3), SD값이 가장 낮은 유전자를 높은 안정성을 가진다고 간주한다. 긴꼬리닭의 심장 조직에서는 RPL13, GUSB, GAPDH가가장 높은 발현 안정성을 나타내고, ACTB, HPRT1, B2M은 낮은 안정성을 보였다. 다리 근육은 HPRT1, GAPDH, RPL13 유전자가 안정성이 높게 나타나며, ACTB, GUSB, EEF1A1은 낮은 안정성을 보인다. 그리고 가슴살에서는 GAPDH, RPL19, HPRT1이 안정성이 높으며, B2M, ACTB, VIM은 안정성이 낮게 나타났다. 이 결과를 종합해 보면 GAPDH는 모든 조직에서 안정성이 높은 유전자로 선택되었다(Fig. 4).
Table 3.
Bestkeeper software statistical analysis of housekeeping gene in each tissues of long-tailed chicken
| (A) Bestkeeper analysis results in heart tissues |
|
|
B2M
|
SDHA
|
GAPDH
|
RPL13
|
VIM
|
EEF1A1
|
HPRT1
|
GUSB
|
RPL19
|
ACTB
|
ABL1
|
|
N
|
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
|
GM [Ct]
|
19.47 |
23.67 |
19.26 |
23.19 |
24.09 |
19.97 |
23.33 |
27.27 |
23.32 |
20.16 |
25.13 |
|
AM [Ct]
|
19.50 |
23.69 |
19.27 |
23.19 |
24.10 |
19.98 |
23.34 |
27.27 |
23.34 |
20.18 |
25.13 |
|
Min [Ct]
|
18.24 |
22.92 |
18.79 |
22.91 |
23.50 |
19.31 |
22.63 |
26.90 |
22.73 |
19.08 |
24.52 |
|
Max [Ct]
|
20.94 |
24.63 |
19.97 |
23.46 |
24.86 |
20.68 |
24.50 |
27.73 |
24.37 |
20.97 |
25.97 |
|
SD [± Ct]
|
0.96
|
0.63
|
0.46
|
0.19
|
0.51
|
0.47
|
0.77
|
0.31
|
0.69
|
0.73
|
0.56
|
|
CV [% Ct]
|
4.90 |
2.66 |
2.41 |
0.82 |
2.12 |
2.36 |
3.30 |
1.12 |
2.95 |
3.63 |
2.22 |
|
Max [x-fold]
|
–2.35 |
–1.68 |
–1.39 |
–1.22 |
–1.51 |
–1.58 |
–1.63 |
–1.29 |
–1.51 |
–2.12 |
–1.52 |
|
Max [x-fold]
|
2.76 |
1.94 |
1.63 |
1.20 |
1.71 |
1.64 |
2.25 |
1.38 |
2.06 |
1.75 |
1.79 |
|
SD [± x-fold]
|
1.94 |
1.55 |
1.38 |
1.14 |
1.42 |
1.39 |
1.71 |
1.24 |
1.61 |
1.66 |
1.47 |
| (B) Bestkeeper analysis results in thigh tissues |
|
|
B2M
|
SDHA
|
GAPDH
|
RPL13
|
VIM
|
EEF1A1
|
HPRT1
|
GUSB
|
RPL19
|
ACTB
|
ABL1
|
|
N
|
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
|
GM [Ct]
|
19.87 |
25.13 |
16.47 |
27.02 |
23.23 |
20.06 |
24.02 |
26.78 |
21.80 |
19.75 |
27.36 |
|
AM [Ct]
|
19.88 |
25.14 |
16.47 |
27.02 |
23.23 |
20.08 |
24.02 |
26.80 |
21.81 |
19.77 |
27.37 |
|
Min [Ct]
|
18.98 |
24.63 |
15.98 |
26.57 |
22.95 |
18.91 |
23.83 |
26.18 |
20.92 |
18.95 |
26.55 |
|
Max [Ct]
|
20.79 |
25.87 |
16.73 |
27.54 |
23.79 |
20.94 |
24.16 |
27.90 |
22.69 |
20.71 |
27.90 |
|
SD [± Ct]
|
0.61
|
0.49
|
0.33
|
0.35
|
0.37
|
0.78
|
0.13
|
0.73
|
0.59
|
0.63
|
0.55
|
|
CV [% Ct]
|
3.05 |
1.94 |
2.00 |
1.28 |
1.60 |
3.89 |
0.52 |
2.74 |
2.72 |
3.18 |
2.00 |
|
Max [x-fold]
|
–1.85 |
–1.42 |
–1.41 |
–1.36 |
–1.22 |
–2.22 |
–1.14 |
–1.52 |
–1.84 |
–1.75 |
–1.76 |
|
Max [x-fold]
|
1.90 |
1.66 |
1.20 |
1.43 |
1.48 |
1.84 |
1.10 |
2.16 |
1.86 |
1.94 |
1.45 |
|
SD [± x-fold]
|
1.52 |
1.40 |
1.26 |
1.27 |
1.29 |
1.72 |
1.09 |
1.66 |
1.51 |
1.55 |
1.46 |
| (C) Bestkeeper analysis results in breast tissues |
|
|
B2M
|
SDHA
|
GAPDH
|
RPL13
|
VIM
|
EEF1A1
|
HPRT1
|
GUSB
|
RPL19
|
ACTB
|
ABL1
|
|
N
|
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
3 |
|
GM [Ct]
|
20.43 |
24.28 |
15.25 |
25.30 |
23.14 |
20.39 |
23.35 |
26.07 |
22.13 |
19.43 |
27.07 |
|
AM [Ct]
|
20.44 |
24.29 |
15.26 |
25.31 |
23.15 |
20.40 |
23.36 |
26.08 |
22.13 |
19.44 |
27.07 |
|
Min [Ct]
|
19.57 |
23.78 |
14.95 |
24.93 |
22.20 |
19.61 |
22.90 |
25.42 |
21.92 |
18.97 |
26.60 |
|
Max [Ct]
|
20.94 |
24.92 |
15.47 |
26.06 |
23.92 |
20.96 |
23.92 |
26.92 |
22.52 |
20.38 |
27.71 |
|
SD [± Ct]
|
0.58
|
0.42
|
0.20
|
0.50
|
0.63
|
0.52
|
0.38
|
0.56
|
0.26
|
0.63
|
0.42
|
|
CV [% Ct]
|
2.83 |
1.74 |
1.34 |
1.98 |
2.73 |
2.56 |
1.61 |
2.15 |
1.17 |
3.22 |
1.56 |
|
Max [x-fold]
|
–1.81 |
–1.42 |
–1.24 |
–1.29 |
–1.92 |
–1.71 |
–1.37 |
–1.57 |
–1.16 |
–1.38 |
–1.39 |
|
Max [x-fold]
|
1.42 |
1.55 |
1.16 |
1.69 |
1.71 |
1.48 |
1.48 |
1.80 |
1.31 |
1.93 |
1.56 |
|
SD [± x-fold]
|
1.49 |
1.34 |
1.15 |
1.41 |
1.55 |
1.44 |
1.30 |
1.48 |
1.20 |
1.54 |
1.34 |
Download Excel Table
Fig. 4.
Standard deviation value of 11 housekeeping genes determined by bestkeeper analysis in (A) heart, (B) thigh, and (C) breast of long-tailed chicken. Low standard deviation values indicate stable expression.
Download Original Figure
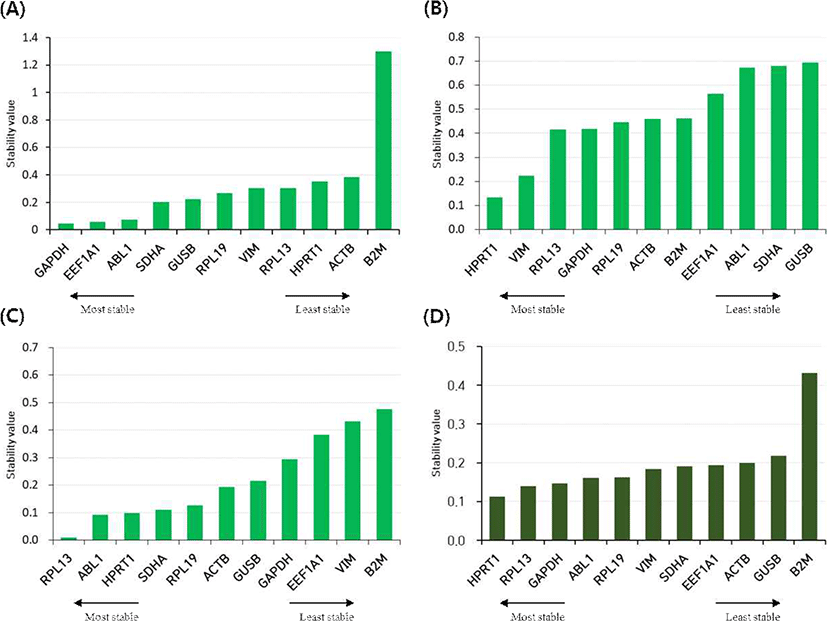
NormFinder 분석
모든 하우스키핑 유전자의 전체 발현 변이를 추정하여 안정성 값을 계산하는 NormFinder 분석은 심장 조직에서 GAPDH, EEF1A1, ABL1이 높은 안정성의 유전자로 선택되었고, HPRT1, ACTB, B2M이 낮은 안정성의 유전자로 선별되었으며, 다리 근육은 HPRT1, VIM, RPL13이 높은 안정성, ABL1, SDHA, GUSB가 낮은 안정성을 보였다. 가슴살 조직은 RPL13, ABL1, HPRT1의 순으로 안정성 점수가 높게 나타났고, EEF1A1, VIM, B2M이 낮은 안정성 점수를 나타냈다. 각 조직을 그룹별로 나누어 그룹 간 비교를 통해 유전자의 발현 안정성을 추정해 보았을 때, HPRT1과 RPL13의 조합이 모든 조직에서 안정성이 높은 최적의 기준 유전자로 선택되었다(Fig. 5).
Fig. 5.
Stability values of 11 housekeeping genes analyzed by NormFinder. (A) heart, (B) thigh, (C) breast, and (D) total.
Download Original Figure
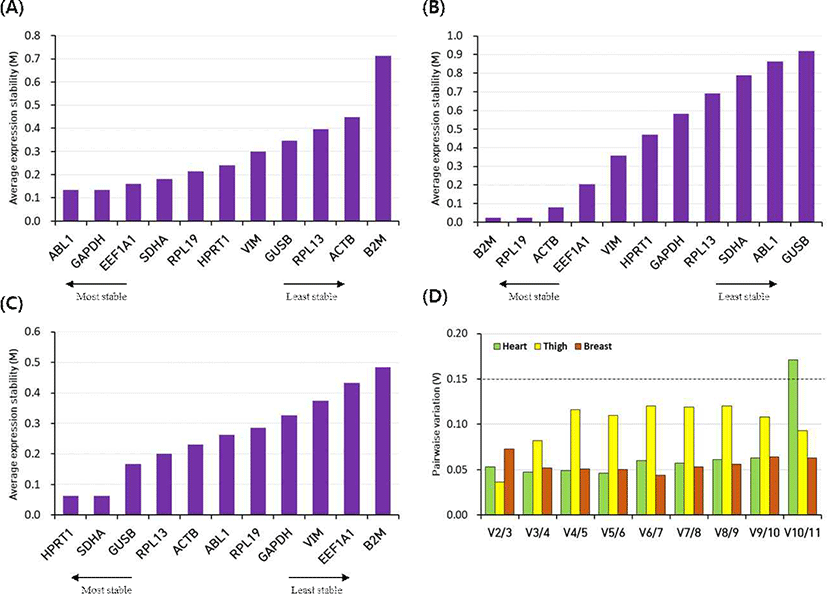
geNorm 분석
geNorm소프트웨어는 M값이 낮을수록 안정성이 높음을 나타내며, 기본값을 M < 1.5로 제한한다[17]. 각 조직에 대한 모든 유전자의 발현 안정성 M값은 모두 1.5 이하로 나타났다. 심장에서 ABL1, GAPDH, EEF1A1가 발현 안정성이 높으며, RPL13, ACTB, B2M이 낮은 발현 안정성을 보였고, 다리 근육에서 B2M, RPL19, ACTB는 안정성이 높으며, SDHA, ABL1, GUSB가 낮은 안정성을 보였다. 가슴살에서 높은 발현 안정성을 가진 유전자는 HPRT1, SDHA, GUSB이며, VIM, EEF1A1, B2M이 낮은 안정성을 나타냈다. 기준 유전자 수를 결정하기 위해 정규화 인자(normalization factor, NF) NFn과 NFn+1 사이의 쌍별 변이(Vn/Vn+1)의 값을 계산하여, 0.15 미만의 값(V < 0.15)으로 제한한다[18]. 이 연구에서 모든 조직의 V 2/3의 값이 0.15 미만이었으며(심장: 0.053, 다리 근육: 0.036, 가슴살: 0.073), 이는 2개 기준 유전자 조합이 데이터의 정규화를 위해 적합하다(Fig. 6).
Fig. 6.
Stability (M) value of 11 housekeeping genes calculated by geNorm analysis in (A) heart, (B) thigh, and (C) breast of long-tailed chicken, (D) pairwise variations (V) value calculated in geNorm analysis to determine the number of reference genes for normalization.
Download Original Figure
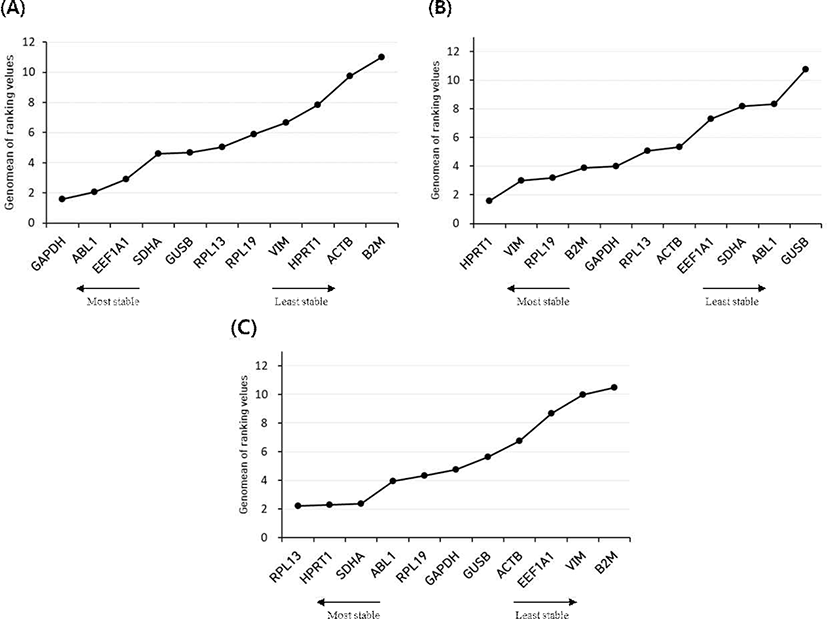
RefFidner 분석
RefFinder는 각 소프트웨어(Bestkeeper, geNorm, NormFiunder)를 통합하여 나온 데이터를 포괄적으로 분석하고, 순위를 기하학적 평균으로 계산해 전체 최종 순위를 정한다(Table 4). 심장 조직은 GAPDH, ABL1, EEF1A1이 가장 높은 안정성을 보였으며, 다리 근육은 HPRT1, VIM, RPL19 그리고 가슴살은 RPL13, HPRT1, SDHA가 안정성이 높은 유전자로 보였다. 낮은 안정성을 가진 유전자는 심장에서 HPRT1, ACTB, B2M이 나타났고, 다리 근육은 SDHA, ABL1, GUSB, 가슴살은 EEF1A1, VIM, B2M이 낮은 안정성을 보였다. HPRT1은 가슴살과 다리 근육에서 안정성이 높지만, 심장에서는 낮은 안정성을 보였으며, B2M은 심장과 다리 근육에서 낮은 안정성을 나타냈다(Fig. 7).
Table 4.
Ranking of housekeeping genes for all software
| Heart |
Thigh |
Breast |
Total |
| Ranking |
Best-Keeper |
Norm Finder |
ge Norm |
Ref-Finder |
Best-Keeper |
Norm Finder |
ge Norm |
Ref-Finder |
Best-Keeper |
Norm Finder |
ge Norm |
Ref-Finder |
RefFinder |
| 1 |
RPL13
|
GAPDH
|
ABL1
|
GAPDH
|
HPRT1
|
HPRT1
|
B2M
|
HPRT1
|
GAPDH
|
RPL13
|
HPRT1
|
RPL13
|
HPRT1
|
| 2 |
GUSB
|
EEF1A1
|
GAPDH
|
ABL1
|
GAPDH
|
VIM
|
RPL19
|
VIM
|
RPL19
|
ABL1
|
SDHA
|
HPRT1
|
VIM
|
| 3 |
GAPDH
|
ABL1
|
EEF1A1
|
EEF1A1
|
RPL13
|
RPL13
|
ACTB
|
RPL19
|
HPRT1
|
HPRT1
|
GUSB
|
SDHA
|
ACTB
|
| 4 |
EEF1A1
|
SDHA
|
SDHA
|
SDHA
|
VIM
|
GAPDH
|
EEF1A1
|
B2M
|
SDHA
|
SDHA
|
RPL13
|
ABL1
|
EEF1A1
|
| 5 |
VIM
|
GUSB
|
RPL19
|
GUSB
|
SDHA
|
RPL19
|
VIM
|
GAPDH
|
ABL1
|
RPL19
|
ACTB
|
RPL19
|
RPL19
|
| 6 |
ABL1
|
RPL19
|
HPRT1
|
RPL13
|
ABL1
|
ACTB
|
HPRT1
|
RPL13
|
RPL13
|
ACTB
|
ABL1
|
GAPDH
|
GUSB
|
| 7 |
SDHA
|
VIM
|
VIM
|
RPL19
|
RPL19
|
B2M
|
GAPDH
|
ACTB
|
EEF1A1
|
GUSB
|
RPL19
|
GUSB
|
SDHA
|
| 8 |
RPL19
|
RPL13
|
GUSB
|
VIM
|
B2M
|
EEF1A1
|
RPL13
|
EEF1A1
|
GUSB
|
GAPDH
|
GAPDH
|
ACTB
|
B2M
|
| 9 |
ACTB
|
HPRT1
|
RPL13
|
HPRT1
|
ACTB
|
ABL1
|
SDHA
|
SDHA
|
B2M
|
EEF1A1
|
VIM
|
EEF1A1
|
ABL1
|
| 10 |
HPRT1
|
ACTB
|
ACTB
|
ACTB
|
GUSB
|
SDHA
|
ABL1
|
ABL1
|
ACTB
|
VIM
|
EEF1A1
|
VIM
|
RPL13
|
| 11 |
B2M
|
B2M
|
B2M
|
B2M
|
EEF1A1
|
GUSB
|
GUSB
|
GUSB
|
VIM
|
B2M
|
B2M
|
B2M
|
GAPDH
|
Download Excel Table
Fig. 7.
Expression stability ranking of the housekeeping genes calculated by the RefFinder. (A) heart, (B) thigh, and (C) breast.
Download Original Figure
Discussion
연구의 목적에 적합한 기준 유전자 세트는 실험대상 조직(본 연구에서 골격근으로 구성된 심장, 다리근육, 가슴살)에서 하우스키핑 유전자로서 발현이 높아 쉽게 측정이 가능하여야 하고, 발현 안정성을 유지하는 것과 더불어, 동일 실험조건에서 다른 조직(평활근으로 구성된 소장, 대장 등)에서도 발현 안정성이 유지되어야 한다.
유전자 발현 연구에서 RT-qPCR분석은 mRNA의 발현 수준을 정규화하기 위해 적절한 기준 유전자가 필요하다. 대부분의 RT-qPCR실험에서 기준 유전자로 많이 사용되는 GAPDH, ACTB 및 HPRT1등과 같은 하우스키핑 유전자들이 세포의 기본적인 기능을 담당하기 때문에, 다양한 조건에서도 안정적인 발현을 나타낼 것이라고 생각한다. 하지만 GAPDH는 닭의 가슴 근육 및 인슐린이 자극된 지방세포, ACTB는 저 산소증이 유도된 교모세포종 세포에서 발현이 조절되는 등 특정 실험 조건, 세포·조직 유형 및 대사 과정에 따라 영향을 받기 때문에[19–21], 공통으로 많이 사용되는 하우스키핑 유전자 또한 다양한 조건에서 확인 실험이 필요하다. RT-qPCR에서 적절한 하우스키핑 유전자를 선택하는 것은 유전자 발현 분석에서 기준이 되는 유전자를 정하는 중요한 단계이며, 안정적으로 발현하는 기준 유전자를 선택하여 목적 유전자 발현의 표준화를 통해 실험 결과를 향상시킬 수 있다.
따라서, 본 연구는 긴꼬리닭의 조직(심장, 다리 근육, 가슴살)별로 RT-qPCR데이터를 정규화할 수 있는 기준 유전자를 선별하기 위해 4가지의 분석 소프트웨어 도구(BestKeeper, NormFinder, geNorm 및 RefFinder)에서 11개 하우스키핑 유전자에 대한 발현 안정성을 평가하였다. 분석에 사용한 하우스키핑 유전자는 MHC(major histocompatibility complex)-I 분자의 구성 요소(B2M), 구연산 회로에 관여(SDHA), 당 분해로 인한 에너지 생성에 관여(GAPDH), 리보솜 단백질(RPL13, RPL19), 세포질에서 세포 기관의 위치 결정에 관여(VIM), 번역 인자(EEF1A1), 퓨린 회수 경로에 관여(HPRT1), 리소좀 효소의 구성원(GUSB), 세포 운동성 및 골격과 구조에 관여(ACTB), 세포의 많은 과정에 관여하는 단백질에 생산 관여(ABL1) [22–28]와 같은 각 기관에서 중요한 기능을 담당한다.
4가지 소프트웨어에서 심장 조직은 GAPDH, ABL1 및 EEF1A1을 안정적인 유전자로 선택하였으며, B2M은 모든 소프트웨어에서 가장 낮은 안정성 점수를 받았다. GAPDH의 경우 모든 소프트웨어에서 상위권의 안정성 순위를 나타냈으며, 우리가 연구한 긴꼬리닭의 심장을 포함하여, 인간이나 거위의 심장 조직에서도 안정적인 발현을 나타내 최적의 기준 유전자로 선택되었다[29, 30]. 또한 ABL1은 BestKeeper를 제외한 소프트웨어에서 높은 안정성 순위를 나타냈다. 다리 근육과 가슴살은 HPRT1 유전자를 각각 1순위와 2순위의 높은 안정성을 나타냈으며, 그 외에도 다리 근육에서 VIM과 RPL19 그리고 가슴살에서 RPL13과 SDHA 순으로 가장 안정적인 유전자로 선택되었다.
결론적으로 긴꼬리닭의 조직에서 선택된 참조 유전자는 GAPDH(심장), HPRT1(다리 근육), RPL13(가슴살)이 가장 적합하며, 모든 조직에서는 HPRT1유전자가 가장 안정적인 기준 유전자로 선택되었다. 우리의 연구는 RT-qPCR를 이용한 유전자 발현 연구에서 기준 유전자들의 발현 안정성을 평가함으로써, 참조 유전자 검증의 중요성을 보여준다. 또한 긴꼬리닭을 이용한 향후 유전자 발현 연구에서 다양한 실험 조건과 조직 유형 등에 따라 가장 안정적인 기준 유전자를 확인하는 데 적용될 수 있다.
Acknowledgements
본 논문은 농촌진흥청 연구사업(세부과제번호: PJ01334101, “긴꼬리닭의 유전체 해독 및 유전자 발현 변이정보 지도 구축”)의 지원에 의해 이루어진 것입니다. 또한 본 연구는 2020년도 농촌진흥청 국립축산과학원 전문연구원 과정 지원 사업에 의해 이루어진 것입니다.
References
Zhang J, Gao YY, Huang YQ, Fan Q, Tao X, Wang CK. Selection of housekeeping genes for quantitative gene expression analysis in yellow-feathered broilers. Ital J Anim Sci 2018;17:540-546.

Nam YK, Lee SY, Kim EJ. Evaluation of candidate housekeeping genes for the normalization of RT-qPCR analysis using development embryos and prolarvae in Russian sturgeon Acipenser gueldenstaedtii. Korean J Fish Aquat Sci 2018;51:95-106.
Jee YM. A study on the history of Bong-Whang and Ginkkoridak (a long-tailed cock). AJMAHS 2016;6:391-401.

Borowska D, Rothwell L, Bailey RA, Watson K, Kaiser P. Identification of stable reference genes for quantitative PCR in cells derived from chicken lymphoid organs. Vet Immunol Immunopathol 2016;170:20-24.


de Boever S, Vangestel C, de Backer P, Croubels S, Sys SU. Identification and validation of housekeeping genes as internal control for gene expression in an intravenous LPS inflammation model in chickens. Vet Immunol Immunopathol 2008;122:312-317.


Bagés S, Estany J, Tor M, Pena RN. Investigating reference genes for quantitative real-time PCR analysis across four chicken tissues. Gene 2015;561:82-87.


Pfaffi MW, Tichopad A, Prgomet C, Neuvians TP. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper - Excel based tool using pair-wise correlations. Biotechnol Lett 2004;26: 509-515.


Andersen CL, Jensen JL, Ørntoft TF. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res 2004;64:5245-5250.


Li Y, Han J, Wu J, Li D, Yang X, Huang A, Bu G, Meng F, Kong F, Cao X, Han X, Pan X, Yang S, Zeng X, Du X. Transcriptome-based evaluation and validation of suitable housekeeping gene for quantification real-time PCR under specific experiment condition in teleost fishes. Fish Shellfish Immunol 2020;98:218-223.


Ali H, Du Z, Li X, Yang Q, Zhang YC, Wu M, Li Y, Zhang G. Identification of suitable reference genes for gene expression studies using quantitative polymerase chain reaction in lung cancer in vitro. Mol Med Rep 2015;11:3767-3773.


Adeola F. Normalization of gene expression by quantitative RT-PCR in human cell line: comparison of 12 endogenous reference genes. Ethiop J Health Sci 2018;28:741-748.
Marciano CMM, Ibelli AMG, de Oliveira Peixoto J, Savoldi IR, do Carmo KB, Fernandes LT, Ledur MC. Stable reference genes for expression studies in breast muscle of normal and white striping-affected chickens. Mol Biol Rep 2020;47:45-53.


Sridharan P, Anbazhagan A, Chandrasekaran A, Pratheesh P. Absence of beta-2 microglobulin (B2M) and hypoxanthine-guanine phosphorilbosyl transferase-1 (HPRT1) gene modulation in U87MG and U251 glioblastoma cell lines subjected to cobalt chloride mediated hypoxia. Gene Rep 2018;11: 159-164.

Gill AJ. Succinate dehydrogenase (SDH)-deficient neoplasia. Histopathology 2018;72:106-116.


Caradec J, Sirab N, Keumeugni C, Moutereau S, Chimingqi M, Mater C, Revaud D, Bah M, Manivet P, Conti M, Loric S. ‘Desperate house genes’: the dramatic example of hypoxia. Br J Cancer 2010;102:1037-1043.



Li M, Rao M, Chen K, Zhou J, Song J. Selection of reference genes for gene expression studies in heart failure for left and right ventricles. Gene 2017;620:30-35.