Introduction
인간의 CD24 분자는 1978년 xenogenic rat의 단일클 론항체 생산 시 처음으로 발견되었으며, 열안정성을 보여 heat-stable antigen (HSA)으로도 명명되었다[1]. CD24 는 glycosyl-phospatidylinositol (GPI) anchor에 의해 세포막에 연결되어 있는 구조를 가진 뮤신 타입(mucinetype) 의 세포 부착(cell adhesion) 단백질로[2], 세포 사이 또는 세포외기질 사이의 부착성 상호작용을 촉진하는 기능 을 가진다. 인간 및 마우스의 CD24 분자는 다수의 N-/Oglycosylation site를 가지고 있으며, 이로 인해 글리코실화 (glycosylation) 정도에 따라 다양한 분자량을 갖게 되며 리 간드(ligand) 특이성도 달라진다[1]. 인간의 CD24 분자는 B 림프구(B lymphocyte), 과립구(granulocyte), 상피세포 (epithelial cell), 수지상세포, 신경 모세포(neuroblast) 등 다양한 세포에서 발현되며, 암세포에서는 과발현된다[1]. 특 히 CD24 유전자 발현이 폐암, 난소암, 유방암, 전립선암 및 췌장암 등의 암세포에서 높은 것으로 나타났다[3]. 이러한 특징으로 인해 CD24는 종양 줄기 세포(tumor stem cell) 마커로 활용된다[1]. 일례로 CD24는 CD44 분자와 함께 유 방암 줄기세포를 표지하는 표면 마커로 활용 가능하다[4]. CD24와 결합하는 분자들로는 CD171(L1CAM), TAG-1, Contactin 등이 존재하며, 이 결합분자들은 마우스 뇌에서 신경세포의 성장 억제를 유도하는 CD24 분자의 반응을 매개 하였다[1]. 또한 CD24는 P-selectin에 대한 결합분자로도 작용한다. 혈소판 또는 내피세포에서 발현되는 P-selectin 과 암세포에서 발현되는 CD24 분자 간 결합을 통한 분자 적 상호작용이 암세포의 전이(metastasis)와 관련됨이 보 고되었으며[5], 인간의 CD24 유전자와 P-selectin 유전자 간 물리적 상호작용 네트워크가 형성됨이 확인되었다[6]. 뿐 만 아니라, CD24 분자는 후천성 면역반응과 관련하여 공 동자극 (costimulation) 기능을 가지는 것으로 보고되었다 [7]. 마우스의 수지상세포에서 발현되는 CD24가 CD8+ T 세포 활성화를 조절하는 기능을 가지고 있으며, 수지상 세 포 소집단에서의 CD24 유전자의 발현 수준에 따라 항원 미 접촉 (naïve) CD8+ T 세포는 폐-회기 (lung-homing) T effector 세포 또는 기억세포 유사 (central memory-like) T effector 세포로 분화되었음이 보고되었다[7]. 한편 인 간의 CD24 유전자의 다형성(polymorphism)은 다발성 경 화증(multiple sclerosis), 류머티스성 관절염(rheumatoid arthritis), 전신 홍반성 낭창(systemic lupus erythematosus) 등과 같은 자가면역질환과 연관성을 가지는 것으로 보고된 바 있다[1].
본 연구는 돼지에서 미 확립된 CD24 유전자 구조 분석을 실시하여 엑손(exon) 및 인트론(intron) 구조를 밝힌 후, 엑 손 영역에서의 변이를 검증하여 돼지 CD24 유전자 구조에 관한 기초 정보를 제공하고자 하였다. 이 유전자 정보는 향 후 돼지 면역 반응과 관련된 CD24 분자의 기능적 역할을 구 명하는 연구에 기초 정보로 활용 가능할 것으로 기대된다.
Materials and Methods
인간의 CD24 유전자의 mRNA서열 (GenBank accession no. NM_013230.3)을 Query 서열로 하여 돼지의 Expressed sequence tags (ESTs) database에서 Blastn (https://blast.ncbi.nlm.nih.gov/Blast.cgi) search를 수 행하였고, 그 결과 총 178개의 Blast Hit EST 서열 정보 를 확인하였다. 이들 ESTs는 SeqMan(DNASTAR® Lasergene) 을 이용한 assembly 결과 contig를 생성하였 다. 생성된 contig를 Query 서열로 하여 돼지의 cDNAs database에서 Blastn (https://www.ensemble. org/Multi/Tools/Blast) search를 수행하였고, 그 결과 1 번 염색체 73,119,759-73,124,357 위치에 ENSSSCG00000031023 유전자가 확인 (E-value: 0.0) 되었다. 돼지의 ENSSSCG00000031023 유전자와 연관하여 존재 하는 유전자들로 구성된 상동영역을 인간, 마우스 래트 등과 비교 확인한 결과, 돼지에서도 ENSSSCG00000031023 유 전자 주변 영역에 RTN4IP1, QRSL1, C6orf203, BEND3, PDSS2 등 복수의 유전자가 분포하였으며, 인간 등 다른 종 의 CD24 유전자의 homologue인 것으로 확인되었다.
CLC Genomics Workbench 9.5.2 (QIAGEN) 를 이용하 여 인간, 마우스, 래트 등의 CD24 단백질 암호화 서열(coding sequence)과 단백질 서열들이 정렬되었으며, phylogenetic tree가 생성되었다. phylogentic tree는 단백질 서 열정렬 결과로부터 neighbor joining algorithm을 이용하 여 생성하였다[8]. Distance는 Jukes-Cantor model을 이 용하여 측정하였으며[9], bootstrap test를 10,000 replicates로 실시하였다.
돼지, 인간, 소, 양, 마우스, 래트의 CD24 단백질에 대 한 N 말단 signal peptide, C 말단 GPI-anchoring signal peptide, O-glycosylation site 예측에는 각각 SignalP 4.1 server[10], GPI-SOM program[11], NetOGlyc 4.0 server를 이용하였다[12].
돼지 CD24 단백질 구조는 ab initio 예측을 통해 생성되었 다. 돼지 CD24 단백질은 구성 원자들 간의 상호작용 에너지 를 기술하여 열역학적으로 자유에너지의 최저점(local minimum) 의 구조를 예측하였다. 결합분자인 CD62P(SELP) 단백질 구조는 homology modeling을 적용하여 생성되었 다. 돼지의 CD62P 단백질 서열 정보를 이용하여 2차구조와 도메인 구조 분석을 실시하였다. 이후 다른 종의 CD62P 단 백질 구조에 예측하고자 하는 돼지 CD62P 단백질 서열을 정 렬 시킨 후, side chain이나 loop 등 단백질 구조의 기본 특성 에 대한 에너지 함수를 사용하여 구조를 예측하였다.
추정된 돼지 CD24 유전자의 5’ upstream 지역 2000 bp 서열에 대한 CpG island search 수행을 위해 EMBL-EBI의 EMBOSS Cpgplot tool을 이용하였다[13]. Cpgplot 분석 을 위한 parameter는 window size = 100, minimu length of an island = 200, minimum observe/expected ratio = 0.6, minimum percentage of G+C = 50으로 설정되었다.
국립축산과학원에서 보유중인 4품종(Yorkshire, Landrace, Berkshire, Jeju Black Pig) 및 멧돼지(Wild Boar) 등 총 59두의 genomic DNA 시료(Table 1)를 이용하여 CD24 유전자의 엑손 1번 및 엑손 2번 영역을 증폭시키기 위 하여 중합효소연쇄반응(polymerase chain reaction, PCR) 을 실시하였다. 사용된 primer 서열들은 Table 2에 명시 하였다. Primer3Plus (https://primer3plus.com)를 이용 한 primer 디자인에 ENSSSCG00000031023 서열을 이용 하였다. PCR 반응액은 DNA 50 ng 이상, forward primer/ reverse primer 각각 10 pmol/㎕, dNTP 각 2.5 mM, HS Taq polymerase 2.5 units/㎕, 멸균증류수를 넣어 총 vol-ume을 20 ㎕로 맞추었다. PCR 반응은 95℃에서 initial denaturation( 10 min) 1회 실시된 후, 95℃ denaturation(30 sec), 62℃(엑손1번)/58℃(엑손2번) annealing(30 sec), 72℃ elongation(30 sec) 반응을 총 35회 실시되었으며, 72℃ final elongation(10 min) 1회 실시되었다.
| Breeds | Exon1 | Exon2 |
|---|---|---|
| Yorkshire | 18 | 18 |
| Landrace | 12 | 12 |
| Berkshire | 25 | 25 |
| Jeju Black Pig | 2 | 2 |
| Wild Boar | 2 | 2 |
|
|
||
| total | 59 | 59 |
PCR 산물들은 MultiScreen® HTS PCR plates을 사용하 여 정제되었다. Sequencing 반응액은 BigDye® Terminator Cycle Sequencing Kit를 이용하여 DNA 시료 당 정제 된 PCR 산물 3 ㎕, 5x Buffer 1.75 ㎕, Big dye 0.5 ㎕, forward/reverse primer(1.6 pmol/㎕) 1 ㎕, 멸균 증류수 3.75 ㎕로 맞추어 총 volume이 10 ㎕가 되게 하였다. Sequencing 반응은 94℃ 10초간 변성, 60℃에서 4분간 annealing 과정을 35번 반복하였다. 반응 산물들은 isopropanol/ ethanol 침전 방식으로 정제된 후 ABI 3730/3730xl DNA Analyzer를 사용하여 모세관 전기영동을 통해 서열을 결정하였다. 얻어진 염기서열은 SeqMan(DNASTAR Lasergene) 을 사용하여 조립하고, 품종별 변이(variant) 분석 에 이용되었다.
Results and Discussion
ENSSSCG00000031023 전사체 (ENSSSCT00000036499.2) 서열에 대한 BLASTn (https:// blast.ncbi.nlm.nih.gov/Blast.cgi) 분석을 수행한 결과, 염 색체상의 동일 위치에 non-coding RNA(GenBank accession no. XR_002343684)와 일치하였다(일치도: 100 %). SeqMan을 이용한 ENSSSCT00000036499.2 서 열 및 XR_002343684 서열에 대한 분석 결과, ENSSSCT00000036499.2을 contig로 XR_002343684 서열이 100% 일치하였으며, ENSSSCT00000036499.2 coding sequence와도 100% 일치되었다. SeqMan을 이용한 인간 CD24 유전자의 non-coding transcript(NR_117089.1 및 NR_117090.1)와 단백질 코딩 유전자 transcript(NM_001291737.1 및 NM_013230.3) 서열 과 비교 분석 결과, non-coding transcript는 proteincoding transcript 서열에서 internal exon이 빠진 구조였 다. 인간에서와 같이 돼지의 경우 XR_002343684 서열이 non-coding RNA 유전자 구조를 보이지 않고 ENSSSCT00000036499.2 서열과 일치하여 XR_002343684 서 열이 annotation error를 갖고 있는 것으로 사료된다.
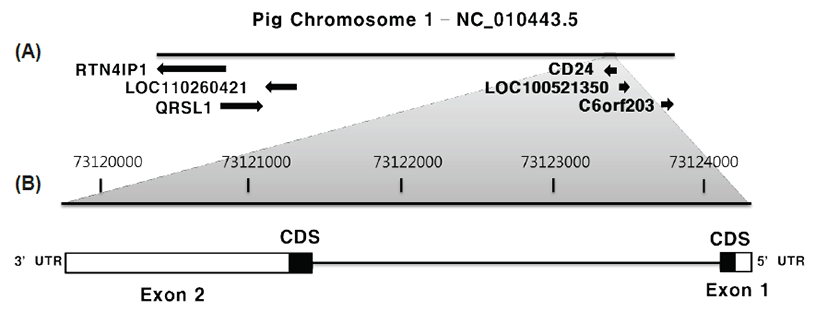
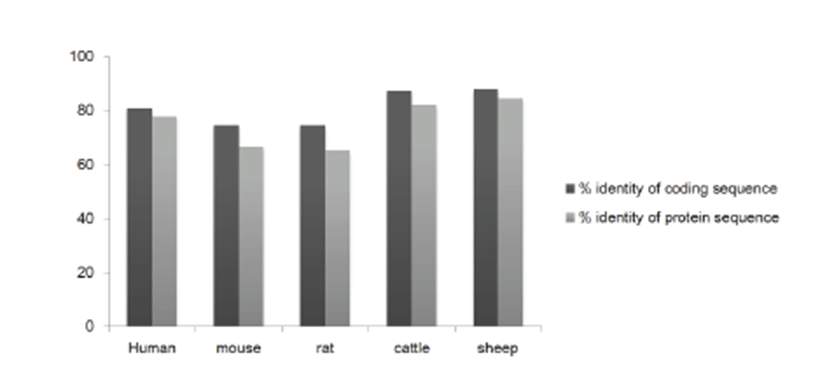
ENSSSCG00000031023 유전자의 transcript(EN SSSCT00000036499.2) 서열에 대한 Blast 분석결 과 인간(NM_013230.3), 마우스(NM_009846.2), 래 트(NM_012752.3)의 CD24 mRNA와 각각 80%, 88%, 83%의 동일성을 가지는 것으로 확인되었다. 이외에 도 소(XM_002690126.3), 말(XM_014735266.1), 양 (XM_004011234.3), 마카크원숭이(XM_005552236.2) 와는 각각 88%, 85%, 86%, 86%로 mRNA 서열이 일치 하였다. 따라서 Ensemble 및 NCBI genome database 에 대한 BLAST 분석 결과, 돼지 유전체 상의 ENSSSCG00000031023 유전자 주변 영역 유전자들의 분 포 및 synteny, 인간·마우스·래트 등 다양한 종과의 CD24 mRNA 서열 유사성를 근거로 돼지에서 ENSSSCG00000031023 유전자를 인간의 CD24 유전자와 상동인 단백질 암호화 유전자로 추정하였다(Fig 1)[14].
돼지의 CD24 유전자(ENSSSCG00000031023)에 대한 Gene expression databases(Bgee, Genvisible) search 결과, 정상 생리 조건의 돼지에서 전전두엽 피질, 심장, 폐, 간, 신장, 갑상선, 소장, 회장·공장·결장 점막 등에서 발현 되는 것이 확인되었으며[15, 16], PEDE (Pig Expression Data Explorer) database[17]에서 두 종의 full-length cDNA library 서열(GenBank accession no. AK235648.1 및 AK392842.1)과 99% 일치하여 돼지에서의 CD24 유전 자 발현을 확인하였다.
돼지의 CD24 유전자는 1번 염색체(reverse strand) 73,119,759-73,124,357번 서열에 위치하고 유전자 길 이는 4599 bp이며 1,841 bp 길이의 전사체(ENSSSCT00000036499.2) 로 전사된다. 이 transcript는 두 개의 엑손(exon) 및 인트론(intron) 한 개로 구성되어 있고 인 트론은 GT/AG rule을 따랐다(Fig 1). 엑손 1번, 엑손 2번, 인트론은 각각 160 bp, 1681 bp, 2758 bp 길이이며, 엑손 2번은 특히, 1513 bp 길이의 긴 3’ UTR(untranslated region)을 가지고 있었다(Table 3). 이러한 구조는 인간, 마 우스, 래트 등의 CD24 유전자 구조와 유사하였다[14, 18].
Transcript 서열에 대하여 ORFinder(https://www. ncbi.nlm.nih.gov/orffinder)를 이용한 ORF(open reading frame) search 결과 총 18개의 ORFs가 발견되었다. ORFs에 대한 Blastp 및 SmartBLAST search 결과 78개의 아미노산 을 암호화하는 237 bp 길이의 짧은 ORF가 인간 뿐만 아니라 다른 종의 CD24 단백질과 각각 정렬되었다. 또한 이 ORF의 아미노산 서열은 transcript(ENSSSCT00000036499.2) 의 번역아미노산인 ENSSSCP00000029101 서열과 100% 일치하였다. 돼지의 CD24 유전자의 번역개시서열은 transcript 서열의 92번째부터 시작되며, 번역종료는 328번째 에서 끝난다.
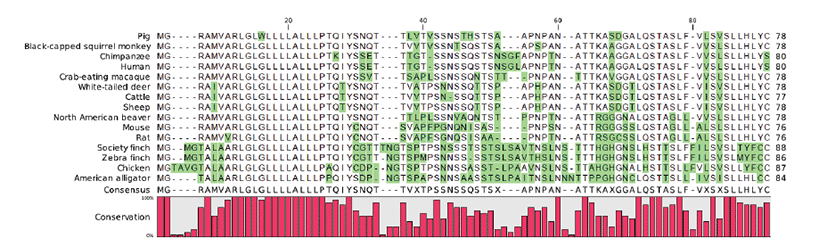
돼지와 인간, 마우스, 래트 및 주요 축종인 소, 양과의 coding sequence 정렬 결과(Fig 2), 돼지의 코딩서열과 각각 80.89%, 74.48%, 74.48%, 87.34%, 88.19%의 동일성을 나타낸다. 6종 모두 coding exon이 2개 이며, 특히 coding exon 1번의 구조가 번역개시 코돈(ATG) 으로부터 총 69 bp 길이의 서열을 가진 coding region으로 구성되 었다. Coding exon 2번은 돼지, 인간, 마우스, 래트에서는 ATTTA로 보존된 서열로 시작되어 각각 168 bp, 174 bp, 162 bp, 162 bp 길이의 coding region으로 구성되었다. 소 및 양에서는 ACTTA로 보존된 서열로 시작된 각 165 bp 및 168 bp 길이의 coding region으로 구성되었다.

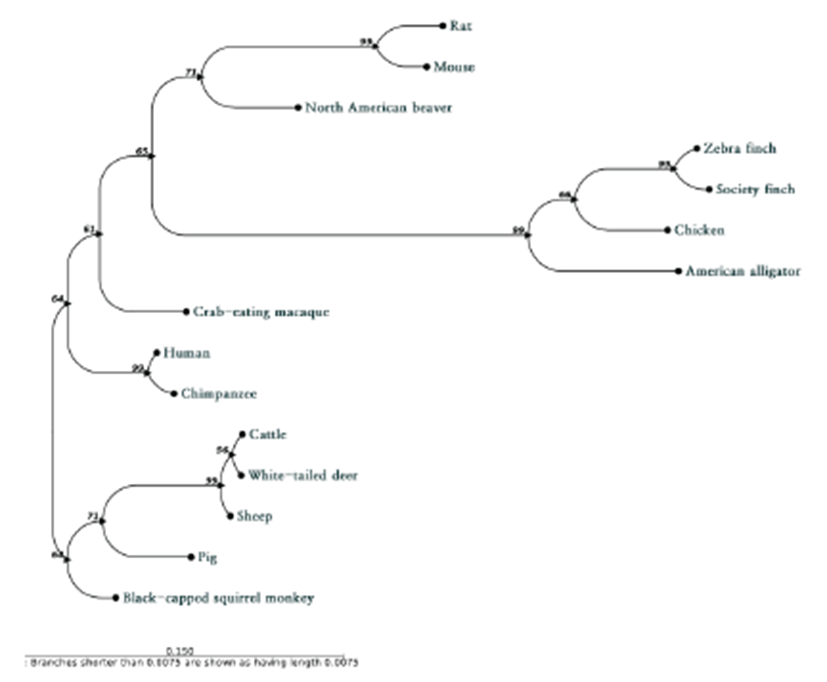
CD24 단백질 서열 정렬 결과(Fig 3), 돼지의 CD24 단 백질은 검은머리 다람쥐 원숭이(Black-capped squirrel monkey)와 87.18%의 가장 높은 동일성을 나타낸다. 또한 인간, 마우스, 래트, 소, 양과 각각 77.78%, 66.67%, 65.38%, 82.05%, 84.62%의 identity를 보여주었다. 닭, 금 화조(Zebra finch), 십자매(Society finch)와 같은 조류와 는 각각 49.44%, 48.28%, 48.86%의 동일성을 나타낸다. Fig 4
CD24 단백질 서열 정렬 결과를 토대로 작성된 phylogenetic tree(Fig 5)는 돼지의 CD24 단백질이 소, 사슴, 양과 같이 반추류(ruminantia)와 진화적 거리가 가장 가까운 것 으로 분석되었으며, 그 다음으로는 검은 머리 다람쥐 원숭 이(Black-capped squirrel monkey)와 가까웠다. 돼지의 CD24와 진화적으로 가장 먼 관계는 금화조(Zebra finch) 및 십자매(Society finch)의 CD24인 것으로 나타났다. 또 한, 돼지의 CD24와 인간의 CD24는 마우스 및 래트의 CD24 보다는 진화적으로 더 가까운 것으로 나타났다.
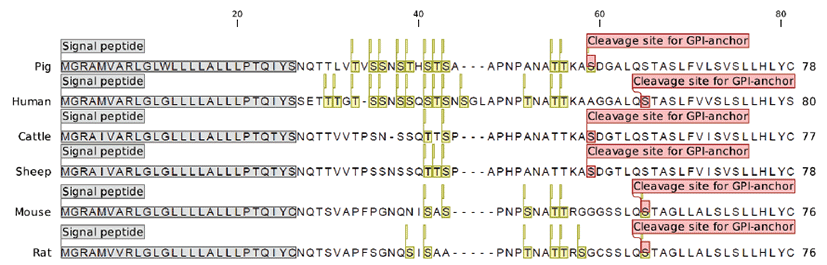
단백질의 C 말단 서열은 번역 후 가공과정을 거쳐 변형될 수 있는데, 가장 일반적인 가공과정이 GPI-anchoring이며, 이 변형은 transmembrane domain 없이 단백질이 세포막 과 연결될 수 있게 한다[19, 20]. 마우스 및 래트에서 실험적 으로 검증된 CD24 단백질도 GPI-anchoring 단백질 중 하 나이며[21], CD24 단백질의 mature form은 번역 서열에서 N 말단 서열의 signal peptide가 잘려나가며 이후 GPI anchoring signal peptide 이후의 C-말단 서열도 잘려나간 형 태를 가진 mucin-like 단백질 구조를 형성한다[22]. 따라서 돼지, 인간, 소, 양, 마우스, 래트의 CD24 단백질에 대한 N 말 단 signal peptide 및 C 말단 GPI-anchoring signal peptide를 인실리코(in silico)로 분석한 결과(Fig 6), 종간 유사 한 형태로 N-말단의 signal, C-말단의 GPI anchoring 신 호가 예측되었다. 돼지 CD24 단백질의 예측 N-말단 signal peptide 영역인 1 - 26번째 아미노산 서열 및 예측 C-말단 GPI-anchoring signal peptide 영역인 57-78번까지의 아 미노산 서열을 다른 종들과 비교한 결과 mature peptide 서 열보다 더 보존된 경향을 보여주었다[23].
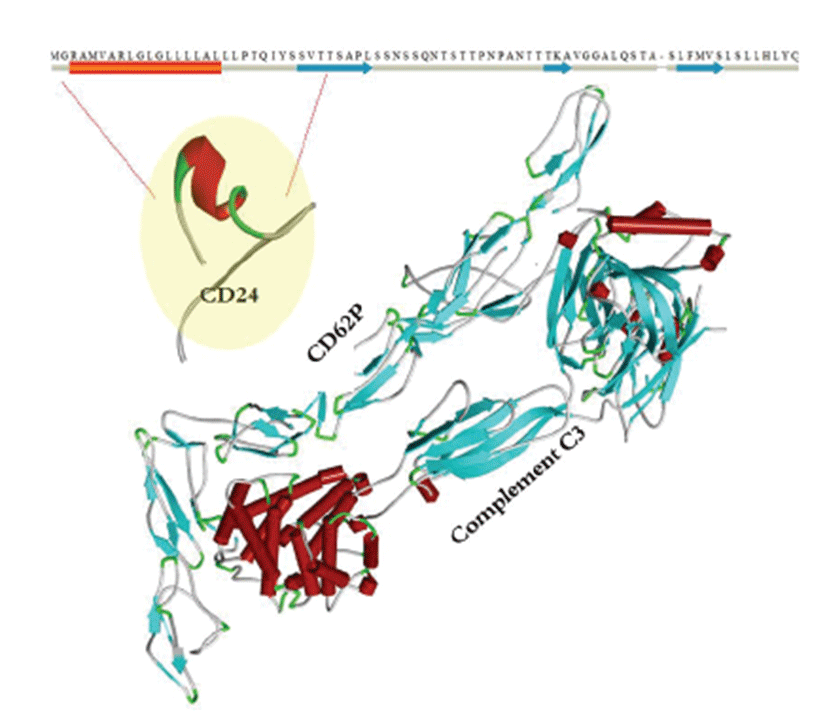
세포부착에 관여하는 CD24 단백질과 보체 C3로 marking 된 CD62P(P-selectin)간의 복합체구조를 모형화하였다 (Fig 7). CD62P와 같은 셀렉틴(selectin), 인테그린(integrin), 케모카인(chemokine) 및 케모카인 수용체 등은 면역 세포의 이동에 관여하는 분자로, 세포부착분자(cell adhesion molecule, CAM)로 분류된다[24]. 셀렉틴은 뮤신-유 사(mucin-like) 단백질과 결합이 가능하며 CD62L(Lselectin), CD62E(E-selectin), CD62P(P-selectin)으 로 나뉜다. 이중에서도 P-selectin은 염증 반응시 백혈구 부착 및 구르기(rolling)를 통한 조직 내로의 이동에 관여한 다[25]. 뮤신-유사 단백질인 CD24를 발현하는 유방암세 포는 혈류 혈소판(platelet)에 결합한 후 혈관내피세포에서 발현하는 P-selectin 분자와 결합을 통한 구르기(rolling) 을 시작하여 암세포가 전이되는 기작이 보고된 바 있다[26]. 보체 단백질 중 하나인 C3는 뇌 염증 발생시 내피세포 활성 화 및 백혈구 포집(recruitment)에 주요한 역할을 하는 것 으로 보고되었다[27]. C3 결핍 마우스의 뇌에서 CD62P, CD62E, ICAM-1, VCAM-1발현이 유의적으로 감소되는 것이 발견되었다[27]. 또한 C3가 절단된 산물인 C3a는 마 우스 뇌 내피세포의 활성화 및 세포 부착분자의 발현을 유도 하는 기능을 하였다[27]. 따라서 돼지에서 P-selectin- CD24-C3 binding pathway 연관 기능을 연구하는 데 위 단백질 복합체 구조가 유용한 기초 정보로 활용될 수 있다.
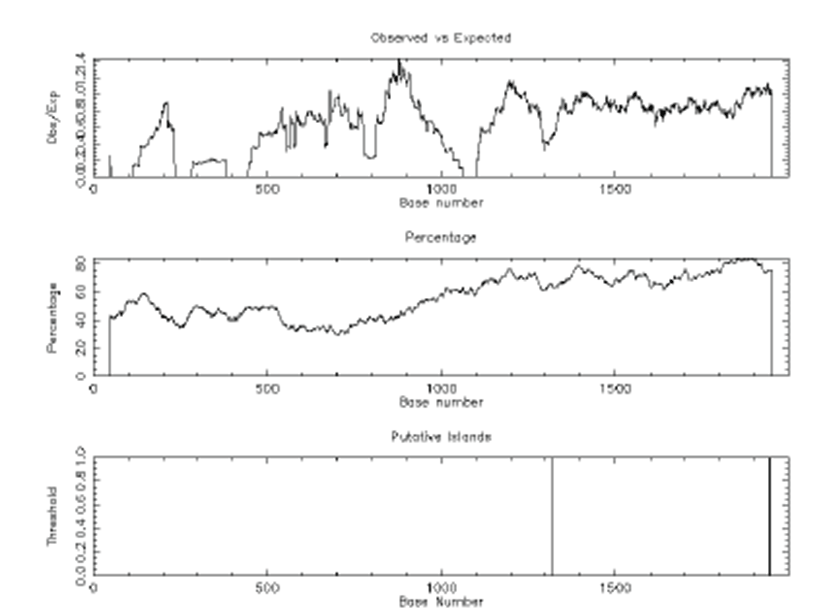
돼지 CD24 유전자의 5’ upstream 지역의 2000 bp 서 열에 대한 조절영역 분석 결과(Fig 8), 624 bp 길이의 CpG island가 예측되었다. CpG island는 유전자 발현 유무를 결 정하는 중요한 조절영역이다[28]. CpG island는 발현되는 유전자에 대한 서열상의 특징이며 일반적으로 전사개시부위 부근에 위치한다[28]. CpG island 내의 CG 비율은 다른 유 전체 영역보다 높은 경향을 보인다[28]. CpG island의 C 염 기가 메틸화(methylation)되면 유전자 발현이 억제되는 반 면 메틸화되지 않으면 발현이 유도된다[28]. CpG plot 분석 결과, 돼지 CD24 유전자가 메틸화에 의해 발현이 조절되는 유전자임을 예측할 수 있었다.
돼지 CD24 유전자는 엑손 1번에서는 변이가 발굴되지 않 았고, 엑손 2번에서 총 11개의 변이가 발굴되었다(Table 4). 이들 변이는 모든 단백질 암호화영역이 아닌 3’ UTR에 서 발굴되었으며, 변이 유형으로 SNP가 8개, 삽입변이가 3 개 발굴되었다(Table 4). 특히 엑손 2번 405-406번 서열 에서의 삽입변이(rs713584359)는 랜드레이스, 멧돼지에 서는 확인되었으나, 요크셔 및 버크셔에서는 확인되지 않았 다. ENSSSCG00000031023 유전자에서 보고된 변이들은 엑손 1번에서는 86번째 서열의 5’ UTR SNP, 157번재 서 열의 동의성 SNP(synonymous SNP) 등 총 2종의 변이가 보고된 바 있고, 엑손 2번에서는 35번째 서열의 stop gained SNP, 51번째 nonsynonymous SNP 등 총 105종의 변이들 이 보고된 바 있다[14]. 하지만, 기 보고된 총 107종의 변이 들 중 11종만이 Direct sequencing을 통해 실제 확인되어 큰 차이를 보여주었다. dbSNP[29]상에 등록된 돼지 SNP 정보는 전 세계적으로 분석된 돼지 개체들로부터 얻은 모든 SNP들이 저장된 것이므로 본 연구의 변이 검증 분석에 포함 되지 않은 듀록, 한국재래돼지, 햄프셔 등의 다양한 품종들의 CD24 엑손 2번 영역에서의 변이 검증이 추가적으로 필요할 것으로 판단된다.
Conclusion
돼지 CD24 유전자는 1번 염색체(reverse strand) 73,119,759-73,124,357번 서열에 위치하고 1,841 bp 길 이의 전사체(ENSSSCT00000036499.2)로 전사된다. 이 전사체는 엑손 2개 및 인트론 1개로 이루어져 있고, 78개 아미노산 서열로 구성된 뮤신 타입(mucine-type)의 세포 부착(cell adhesion) 단백질을 암호화하는 것으로 분석되었 다. CD24는 다양한 기능이 보고되었음에도 불구하고 돼지 에서는 아직까지 기능 연구가 미진한 상황이다. 따라서 돼지 CD24 단백질의 부착기능이 돼지 백혈구 등 면역세포 기능 에 미치는 영향 및 작용 기전에 대한 심층 연구가 필요하며, 돼지에서의 P-selectin-CD24-C3 binding pathway 연관 기능을 밝히는 추가 연구도 필요할 것으로 사료된다. 돼지는 축산 식품으로서의 실용적 가치뿐만 아니라 질환 모델 동물 로서의 잠재력을 보유한 유용 가축이다. 돼지에서의 CD24 유전자 구조 정보는 향후 인간에서 CD24 연관 질병 또는 증 상 연구에 필요한 비교 유전자 정보로 활용될 수 있다. 한편 마우스의 백색 지방 조직 내 CD24를 발현하는 세포 집단이 지방 전구 세포로서 확인된 보고에 의하면[30], 추후 돼지 백 색지방 조직에서의 CD24+ 세포를 이용한 지방 세포 수 조 절과 관련된 기작을 밝히는 데에도 이번 연구결과가 기초 정 보로서 제공될 수 있을 것으로 기대된다. 마지막으로 돼지 특 이적 CD24 항체를 이용하여 CD24 유전자에 대한 세포 수 준에서의 발현 및 기능 검증을 통해 돼지 세포에서의 CD24 의 분자적 특성을 밝히는 연구가 필요할 것으로 판단된다.