ARTICLE
Identification of genomic regions associated with piglet survival and mortality
Yoon-Jung Choi1, Sang-Wook Kim1, Joon-Ho Yoon1, Bong-Hwan Choi2, Tae-Hun Kim2, Kwan-Suk Kim1,*
Author Information & Copyright ▼
1College of Agriculture, Life and Environment Sciences, Chungbuk National University, Cheongju 361-746, Korea
2Division of Animal Genomics and Bioinformatics, National Institute of Animal Science, Suwon 441-706, Korea
*Corresponding Author :Kwan-Suk Kim, College of Agriculture, Life and Environment Sciences, Chungbuk National University, Cheongju 361-746, Korea, Tel: +82-43-261-2547, Fax: +82-43-273-2240 ; E-mail:
kwansukkim@chungbuk.ac.kr
© Research Institute of Veterinary Medicine, Chungbuk National University. All rights reserved. This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Received: May 21, 2014; Revised: Jun 11, 2014; Accepted: Jun 12, 2014
Abstract
One of the major problems in the modern pig industry is infectious disease. Susceptibility to infectious diseases is influenced by both constitutional (e.g. genotype, age, gender, and reproductive status) and environmental factors (e.g. nutrition, management, infections, and other forms of stress). Genetic variations within individual animals or herds can cause differences in the execution of immune functions against infectious agents in domestic pigs. The objectives of the present study were to identify genetic factor(s) responsible for piglet survival and mortality under commercial field conditions with infectious diseases as well as determine QTL regions for immune capacity in a pig reference family. Sex ratio was examined between normal grown and presumed dead groups of F2 animals generated by Korean native pigs (KNP) and Yorkshire (YS) breeds. The ratio of males was significantly higher in the presumed dead group than in the normal grown group. In order to study genetic factors associated with presumed mortality under disease outbreak, allelic frequencies of 239 single nucleotide polymorphisms (SNPs) were compared between normal grown and presumed dead groups. Exactly 22 SNPs showed significant differences in their allelic frequencies between the two groups, and four of them were validated with another commercial population divided by normal and emaciated pigs. Furthermore, the relationships between 239 SNPs and immune-related traits were studied. These results demonstrate that identification of genetic components of animal immune systems and susceptibility to infectious diseases is possible and will be useful to improve disease resistance in individuals as well as in breeding programs.
Keywords: SNPs; piglet survival; piglet mortality; Korean native pig; pig populations
Introduction
국내 양돈업은 생산성 향상이 곧 경쟁력인 가운데 21 세기부터 극심해진 돼지의 전염성 질병으로 인한 양돈산업의 경제적 피해 늘고 있다. 특히, 2010년에는 국내의 구제역(Foot and Mouth Disease, FMD)발생으로 인한 최악의 경제손실이라는 결과를 낳았다. 때문에 소비자들은 악성가축 전염병 및 질병과 식품의 안전성에 대한 관심이 높아지고 있으며, 고품질 및 안전성 지향을 선호하는 시점으로 변화하고 있다.
특히 매년 양돈농가에 많은 피해를 주고 있는 돼지유행성설사병(Porcine Epidemic Diarrhea, PED), 돼지생식기호흡기증후군(Porcine Reproductive and Respiratory Syndrome, PRRS), 그리고 자돈의 이유 후 전신소모성증후군(Postweaning Multisystemic Wasting Syndrome, PMWS)등은 국내에서 가장 많이 발병하는 돼지의 질병이지만, 질병 특성상 백신 사용에도 불구하고 면역효과가 제대로 발휘하지 못하는 경우도 있고, 높은 백신가격으로 돼지 생산비 증가의 원인이 된다[1-4].
개체나 집단별 면역의 기준치의 차이는 감염병원체 요인에 대한 개체나 집단의 유전적 민감성을 반영하기 때문에 질병에 대한 저항성이 뛰어난 개체들을 선발하여 개량에 이용하는 것이 후대의 질병 발병률을 낮추고 생산성을 높일 수 있는 방법이 될 것이다[5].
돼지의 전염병 등 질병에 의한 생산성 저하를 극복하기 위하여 면역관련 형질에 영향을 주는 DNA 마커를 발굴하고 적용하여 질병에 대한 저항성이 우수한 개체 및 종돈을 선발할 수 있는 기술을 개발하기 위해서 선행연구로서 한국재래돼지와 요크셔 품종교배로 생산된 F2자손에서 면역관련 혈액성분에 영향을 주는 염색체 부위를 탐색하여 백혈구 세포의 조성과 면역글로불린 함량에 영향을 주는 DNA마커가 돼지염색체 4, 13, 14, 15번에서 발굴되었다[6].
따라서 본 연구는 면역관련 형질 및 질병에 영향을 줄 수 있을 것으로 사료되는 DNA 마커를 추가적으로 발굴하기 위 해 수행되었으며, 한국재래돼지(Korean native pig, KNP)와 요오크셔(Yorkshire, YS)교배집단에서 생산된 자돈의 생존집단과 폐사집단에서 성별에 따른 생존 및 폐사율에 미치는 영향분석 및 239개의 단일염기변이(single nucleotide polymorphisms, SNPs) 마커들을 이용하여 집단 간의 대립유전자의 빈도를 비교 분석을 수행하였고, 유의적인 빈도차이가 나는 단일염기변이 마커들을 선발하여 상업돈에서 발생한 위축돈 집단과 정상돈 집단에서의 검증분석을 수행하였다.
Materials and Methods
공시재료 및 육질분석
재래돼지(Korean Native pig: KNP) 5마리와 요크셔 (Yorkahire: YS) 9마리를 임의적으로 선발하여 생산된 F1의 자손을 충북 청원군 샘터농장에서 사육하였다. 유전적 다형성을 최대로 발현할 수 있도록 F1 개체선발과 교배를 하였고, 2007년 8월까지 분만된 F2 집단 중 폐사추정돈 268두와 도축된 정상돈 215두를 본 실험에 이용하였다. 또한 검증에 이용된 집단은 A 사업용 종돈회사에서 일반 삼원교잡 비육돈으로 태어난 자손들 중 위축돈 179두와 정상돈 200두 집단을 이용하였다.
총 213두의 꼬리조직 300 mg으로부터 Wizard® Genomic DNA Purification kit (Promega. co., Ltd, USA)를 이용하여 genomic DNA를 분리하여 이용하였다. Genomic DNA 의 분리과정은 꼬리조직 300 mg에 500 μL의 Nuclei Lysis Solution과 0.5 M EDTA solution (pH 8.0) 120 μL를 첨가하고 20 mg/mL의 Proteinase K 17.5 μL를 첨가한 후 55°C에서 하루(overnight)동안 incubation하여 용해시켰다. 그 후 Protein Precipitation Solution 200 μL를 첨가 후 vortexing한 다음 13,000 RPM에서 6분간 원심 분리하였다. 상층액을 isopropanol 600 μL 첨가한 새로운 튜브에 옮겼으며 Inverting 시킨 후 13,000 RPM에서 5분간 원심 분리하였다. 상층액을 버리고 70% ethanol을 600 μL 첨가하여 13,000 RPM에서 5분간 원심 분리한 후 상층액을 버리고 30분간 Dry시켜 genomic DNA를 추출하였다.
DNA 정량 분석은 spectrophotometer (Pharmacia Biotech, England)를 이용하여 260 nm~280 nm에서 흡광도를 측정하여 DNA의 농도와 순도를 확인하였다.
SRY와 ZF (ZFX-ZFY) 유전자의 PCR 기법을 이용한 성감별
폐사추정돈 268두와 정상돈 248두의 꼬리조직에서 추출한 DNA 를 이용하여 Cho (2005) 등[7]이 사용한 SRY 유전자와 ZF (ZFX-ZFY)유전자의 Primer를 이용하여 PCR 분석을 하였다(Table 1).
Table 1.
Primer used for the amplification of porcine SRY and ZF (ZFX-ZFY) genes
| Gene |
Acc. No. |
Primer sequence (5'→3') |
Fragment size(bp) |
| SRY |
U49860 |
CCCTTTTCAAATGGTGCAGT |
704 bp |
| TGTGAGAAAGTCCCGGCTGT |
| ZF |
AF454949 and NM_177491 |
GCTGACCCTGGAGAAGATGA |
476, 379 bp |
| CTTCTTGTTGAGAGTCATTGACAG |
Download Excel Table
DNA 증폭을 위해 사용된 PCR 기계는 PTC-200 themocycler (MJ Research, Watertown MA, USA)이며, DNA 중합효소는 h-Taq polymerase (Solgent, Korea)를 사용하였다. PCR 반응조건은 template DNA 25 ng, primer 0.01 uM, dNTP 5 mM, 10 × PCR buffer 2.5 μL, 그리고 h-Taq DNA polymerase 0.625 units을 넣어 최종 반응액 25 μL을 이용하였다. 반응조건은 최초 95°C에서 15분간 예비가열 한 후 95°C에서 20초 동안 변성시키고, 각 Primer에 대응하는 annealing 온도에서 20초 그리고 72°C에서 30초 합성(extension)시키는 총 40 cycles 반복증폭하고 72°C에서 5분 마지막 합성단계(final extension)를 수행하고 DNA 증폭을 중단하였다. 증폭한 산물들은 10 μL를 취하여 모두 2% agarose gel에서 100 mV 전압에서 20분간 전기영동을 통해 확인하였다.
MassArray Platform을 이용한 단일염기변이 유전자형 분석
239개의 SNP들의 조합을 이용하여 Multiplex PCR primer들과 14개의 Panel을 디자인하였다[9]. Second와 First PCR Primer를 이용하여 Multiplex PCR을 수행하여 증폭한 후 4그룹의 extension primer를 이용하여 MassArray method로 유전자형 분석을 하였다[9, 10].
첫 번째, multiplex-PCR 반응액 조성은 PCR reaction buffer 10 mM (Tris-HCl, pH8.3, 50 mM KCl, 1.5 mM MgCl2)와 2.5 mM dNTPs, 10 pmol first and second primer pairs, 10 ng의 template DNA, 0.5 U h-Taq DNA polymerase (Bioneer co., Ltd)와 Nanopure H2O를 사용하여 총 반응액은 10 μL로 하였다. PCR 반응에는 ABI GeneAmp PCR System 9700 (Perkin-Elmer Co., USA)를 사용하였고, PCR 조건은 95°C에서 15분간 pre-denaturation 한 후 95°C에서 20초간 denatruation, 56°C에서 30초간 annealing, 그리고 72°C에서 1분간 extention을 45cycles 수행한 후 마지막으로 72°C에서 3분간 최종 extention 과정을 수행하였다. PCR 증폭 산물은 증폭된 단편의 크기가 예상된 allele size 범위 내에 존재하는지, PCR 조건의 적정성 여부를 확인하기 위하여 EtBr (ethidium bromide)이 포함된 2% agarose gel에 전기영동하고 UV 상에서 관찰하였다.
두 번째, dNTP 을 제거하기 위해 SAP treatment 과정을 실시하였다. SAP treatment 반응액 조성은 10 × SAP buffer 0.170 μL, SAP enzyme 0.4 U와 Nanopure H2O를 사용하여 총 반응액 2 μL로 하였다. SAP treatment 조건은 37°C에서 20분간 수행 후 85°C에서 5분간 반응을 시켰다.
세 번째, iPLEX 반응을 하기 위해 10 × iPLEX buffer 0.755 μL, iPLEX termination mix 0.200 μL, extension primer 7 uM, iPLEX enzyme 0.0041 μL와 Nanopure H2O를 사용하여 반응액은 2 μL로 하였다. iPLEX 반응조건은 95°C에서 30초간, 95°C에서 5초간, 52°C에서 5초간, 80°C에서 5초간 수행한 후 3 step에서 5 cycles 수행한 후 2 step에서 다시 40 cycle 수행한 후 72°C에서 3분간 과정을 수행하였다. 384 well에서 SpectroCHIP (Sequenom SpectroCHIP®)으로 sample 이동 후 MALDI-TOF mass spectrometry (Sequenom Compact system)를 이용하여 유전자형을 관찰하였으며 Data 분석 및 sorting은 Sequenom 의 SNP 분석 program인 Typer 4.0을 이용하여 genotyping 결과 확인 및 오류 분석 과 Excel program을 이용하여 Data sorting 하였다.
폐사돈과 정상돈 집단에서 단일염기변이 유전자형 빈도 분석
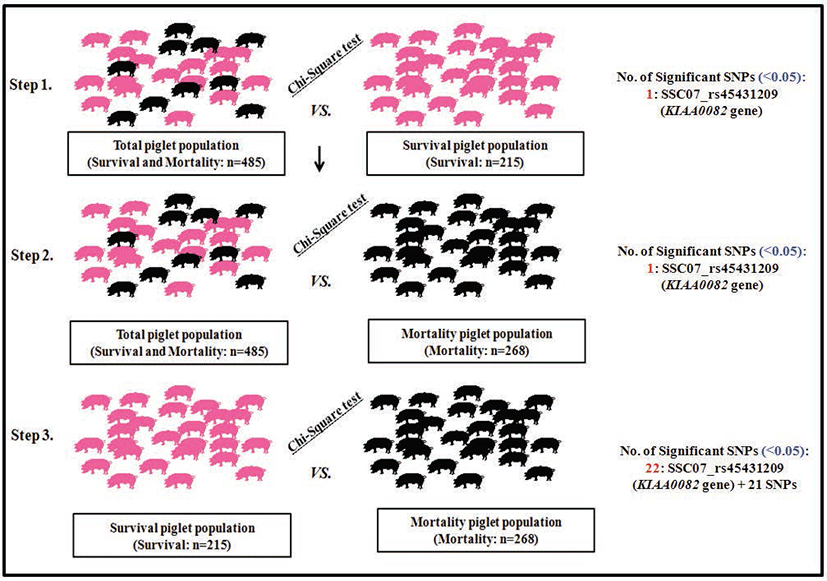
한국재래돼지와 요크셔 교잡돈 F2 집단 485두 중 폐사추정돈 268두와 정상돈 215두로 두 집단을 나누어 239개의 단일염기변이로 3개의 단계별로 빈도 비교를 수행하였다. 첫 번째로 총 485두와 정상돈 215두 집단 간의 대립유전자 빈도를 구해 Chi-Square test를 수행하여 유의적인 효과를 나타내는 마커를 확인하였으며, 두 번째로 총 485두와 폐사추정돈 268두 집단 간의 대립유전자 빈도를 구해 Chi-Square test를 수행여하여 유의적인 마커를 확인하였다. 마지막으로 정상돈 215와 폐사추정돈 268두 집단 간의 대립유전자 빈도를 구해 Chi-Square test를 수행하여 최종 유의적인 마커를 정리 하였다(Fig. 1).
PCR-RFLP method 를 이용한 위축돈과 정상돈 집단에서 검증
PCR-RFLP method를 이용하여 일반 삼원교잡 비육돈으로 태어난 자손 가운데 위축돈 179두와 정상돈 200두 집단에서 검증을 하였다.
돼지 DSP G>A (rs45430738), RASA2 A>C (rs45431691), MYH3 C>A (rs45431421)와 GSN G>A (rs45435111) 단일염기변이들을 포함하는 영역의 증폭을 위해 Oligo 6 (Molecular Biology Insights, Cascade, CO, USA)프로그램을 이용하여 네 개의 Primer를 제작하였다 (Table 2).
Table 2.
PCR primers and restriction enzymes used for genotyping four SNPs
| Gene & SNP |
Reference ID |
Primer sequences (5’→3’) |
Fragment size (bp) |
TA (°C) |
Restriction Enzyme (°C) |
Size (bp) of the allelic polymorphism |
| DSP |
rs45430738 |
CAATGGCGTGACCGATGG |
472 bp |
60°C |
Hpy188I |
307, 263, 165 bp |
| G>A |
TGTGGCTGTGGCGTAGGCT |
(37°C) |
| RASA2 |
rs45431691 |
GTGCTAAAACTGCCTAAG |
102 bp |
54°C |
MseI |
102, 60 bp |
| A>C |
AACCCTAGCACTGTATGG |
(37°C) |
| MYH3 |
rs45431421 |
CGCCGAAAGCTCACTGTG |
265 bp |
55°C |
BslI |
265, 214 bp |
| C>A |
CTGGGGAGGAACCTGCTG |
(55°C) |
| GSN |
rs45435111 |
CGGGAACCTGCAGTATGA |
291 bp |
56°C |
EcoRV |
291, 245 bp |
| G>A |
GGTGGTCTGGTGCGATCA |
(37°C) |
Download Excel Table
DNA 증폭을 위해 사용한 기계는 PTC-200 themocycler (MJ Research, Watertown MA, USA)을 이용하였으며, DNA 중합효소는 h-Taq polymerase (Enzynomics, Korea)를 사용하였다. PCR 반응조건은 주형 DNA 25 ng primer 0.01 uM, dNTP 5 mM, 10 × PCR buffer 2.5 μL, 그리고 h-Taq DNA polymerase 0.625 units을 넣어 최종 반응액 25 μL을 이용하였다.
DSP 유전자 G>A 변이에 대한 유전자형 분석은 증폭한 PCR 산물에서 G 변이를 인식하는 제한효소 Hpy188I을 처리하여 수행하였다. 또한 RASA2 A>C 변이에 대한 유전자형분석은 A 염기를 인식하는 MseI을 처리하여 수행하였으며, MYH3 C>A 변이에 대한 유전자형 분석은 C 염기를 인식하는 BsII을 처리하여 수행하였다. 그리고 GSN 유전자 G>A 변이는 G 염기를 인식하는 제한효소 EcoRV을 처리하여 수행하였다(Table 2). 각각의 변이들에 대한 유전자형 판정은 제한효소 2 unit 을 넣어 각각의 최적반응 조건에서 6시간 이상 반응을 통해 절단된 단편들은 2.5% MetaPhor agarose gel (LONZA, USA)에서 90 mV 전압으로 40분간의 전기영동을 수행한 후 실시하였다.
카이제곱 검증을 이용한 자돈의 성별이 이유 후 폐사율에 미치는 연관성 분석 및 집단과 집단과의 유전자형 빈도분석
카이제곱 검증(Chi-square)은 카테고리 변수 간 차이 혹은 관계를 분석하기 위해서 사용하는 아주 유용한 통계적인 검증 법이다. 관측된 데이터가 예측한 분포를 따르는지 검증하는 방법이며, Golden Helix SNP Variation Suite version 7 (Golden Helix, Inc) software를 이용하여 자돈의 성별이 이유 후 폐사율과 생존율에 연관이 있는지를 검증하기 위해 아래와 같은 생존집단의 유전자형 빈도와 폐사추정집단의 유전자형 빈도간의 차이 역시 아래와 같은 카이제곱 검증법을 이용하였다.
Results and Discussion
SRY 유전자와 ZF (ZFX-ZFY) 유전자의 PCR 기법을 이용한 성감별 결과
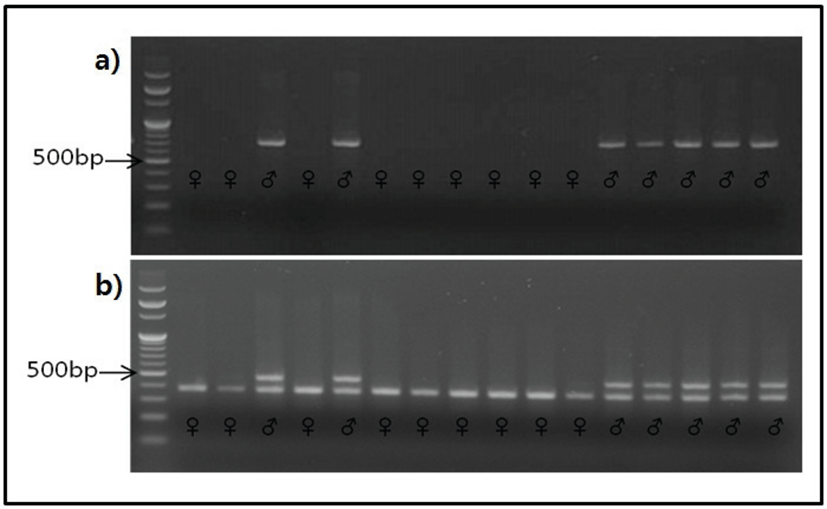
충북 청원군 샘터농장에서 55가계 집단에서 생산된 한국재래돼지와 요크셔 교잡돈 F2 집단 중 폐사추정돈 집단 111두와 정상돈 집단 101두를 제공받아 두 집단의 DNA를 이용하여 PCR 기법으로 정확한 성비를 추정하였다. DNA를 이용한 성감별에는 SRY 유전자를 이용한 방법과, ZF (ZFX-ZFY) 유전자를 이용한 방법 두 가지를 이용하였는데, SRY 유전자는 숫컷(male)일 경우 704 bp에서 증폭이 되며, 암컷(female)일 경우는 증폭이 되지 않는다. 하지만 SRY를 이용한 성감별의 단점은 DNA의 quality 때문에 증폭이 되지 않는 경우가 발생하기 때문에, 시험 상 오류를 범할 수 있다. 따라서 이러한 방법을 보완하기 위해 ZF (ZFX-ZFY) 유전자를 이용한 성감별 방법이 개발되었다[7]. ZF (ZFX-ZFY) 유전자를 이용한 PCR 방법을 이용하였을 경우 숫컷(male)일 경우 476, 379 bp 두개의 증폭 밴드를 확인 할 수 있으며 암컷(female)일 경우는 379 bp 하나에서 증폭 밴드를 확인 할 수 있다(Fig. 2).
폐사돈 및 정상돈 집단에서 성별 분포 및 연관성 결과
정상돈 집단의 성비와 폐사추정돈 집단의 성비의 분포는 Table 3에 나타내었다. 폐사추정돈 집단에서 숫컷(male)의 출현빈도가 높았으며, 정상돈 집단에서는 숫컷보다 암컷(female)의 출현빈도가 높은 것을 관찰 할 수 있었다. 또한 집단별 성비의 빈도차이는 유의적인 효과를 나타내었다 (P<0.05). 본 결과는 선행연구에서 발표된 돼지의 성별이 폐사돈과 정상돈에 유의적인 연관이 있다고 발표한 연구결과와 일치하는 것으로 나타났다[11].
Table 3.
Comparison of sex frequency in Survival and Mortality piglet population
| Sex |
Piglet Survival |
Piglet Mortality |
Total piglet |
Chi-Test (P-value) |
| Male |
0.40 (n=45) |
0.60 (n=67) |
1 (n=112) |
P<0.03 |
| Female |
0.56 (n=57) |
0.44 (n=44) |
1 (n=101) |
P<0.04 |
Download Excel Table
폐사돈과 정상돈 집단에서 유전자형 빈도 분포 및 연관성 결과
한국재래돼지와 요크셔 교잡돈 F2 집단, 폐사추정돈 268두와 정상돈 215두 총 485두를 MassArray method를 이용하여 239개의 단일염기변이의 유전자형을 분석하였다. 집단별 유전자형 빈도 비교를 위해 3단계에 걸쳐 집단간의 유의적인 빈도차이가 나는 마커를 선발하였다(Table 4).
Table 4.
Allele frequencies of 22 SNPs associated with Mortality
| Reference ID |
Allele |
Minor Allele Freq.
|
Chi-Square test 1 |
Chi-Square test 2 |
Chi-Square test 3 |
| Total |
Dead |
Live |
(Total-Dead) |
(Total-Live) |
(Live-Dead) |
| rs45431209 |
A |
0.417 |
0.357 |
0.478 |
0.030 |
0.041 |
0.000 |
| rs45431691 |
A |
0.257 |
0.220 |
0.305 |
0.103 |
0.058 |
0.002 |
| rs45431421 |
C |
0.241 |
0.207 |
0.277 |
0.096 |
0.117 |
0.005 |
| rs45435111 |
A |
0.267 |
0.232 |
0.306 |
0.124 |
0.094 |
0.006 |
| rs45432261 |
C |
0.372 |
0.408 |
0.322 |
0.189 |
0.082 |
0.009 |
| rs45433249 |
A |
0.307 |
0.276 |
0.349 |
0.194 |
0.082 |
0.009 |
| rs10720198 |
A |
0.240 |
0.275 |
0.203 |
0.145 |
0.134 |
0.011 |
| rs45432566 |
T |
0.239 |
0.290 |
0.182 |
0.158 |
0.131 |
0.012 |
| rs45432608 |
C |
0.313 |
0.284 |
0.357 |
0.262 |
0.106 |
0.017 |
| rs45434874 |
C |
0.490 |
0.483 |
0.500 |
0.349 |
0.070 |
0.021 |
| rs45435155 |
T |
0.307 |
0.281 |
0.348 |
0.300 |
0.123 |
0.025 |
| rs45432669 |
A |
0.429 |
0.394 |
0.469 |
0.219 |
0.176 |
0.026 |
| rs45431071 |
C |
0.465 |
0.492 |
0.423 |
0.307 |
0.128 |
0.028 |
| rs10720745 |
A |
0.021 |
0.035 |
0.005 |
0.269 |
0.129 |
0.029 |
| rs45433249 |
C |
0.087 |
0.070 |
0.109 |
0.248 |
0.185 |
0.032 |
| rs45431058 |
G |
0.252 |
0.225 |
0.280 |
0.209 |
0.217 |
0.032 |
| rs45435080 |
G |
0.226 |
0.199 |
0.257 |
0.232 |
0.213 |
0.034 |
| rs45431421 |
C |
0.242 |
0.216 |
0.269 |
0.202 |
0.234 |
0.034 |
| rs45430738 |
G |
0.446 |
0.419 |
0.485 |
0.295 |
0.167 |
0.035 |
| rs10720341 |
C |
0.112 |
0.091 |
0.137 |
0.244 |
0.206 |
0.036 |
| rs45431031 |
C |
0.155 |
0.134 |
0.183 |
0.266 |
0.196 |
0.037 |
| rs45432756 |
G |
0.171 |
0.151 |
0.198 |
0.297 |
0.189 |
0.041 |
Download Excel Table
첫 번째로 총 485두와 정상돈 215두와의 두 집단별 빈도비교를 수행하였으며 유의적인 단일염기변이 마커는 염색체 7번내의 KIAA0082 유전자내에 존재하는 rs45431209 1개로 관찰되었다(P<0.05). 두 번째로 총 485두와 폐사추정돈 268두와의 빈도 비교 결과 역시 첫 번째와 동일한 단일염기변이 마커인 rs45431209 1개로 관찰되었다(P<0.05). 마지막으로 정상돈 215두와 폐사추정돈 268두와의 빈도 비교에서는 총 239개 중 22개의 단일염기변이 마커들이 유의적인 빈도 차이가 있는 것으로 나타났으며, 고도의 유의적인 빈도차이를 보인 마커는 유의적인 22개의 단일염기변이 마커(Table 5) 중 염색체 1번 GSN 유전자내에 존재하는 rs45435111 단일염기변이, 염색체 7번 내 KIAA0082 유전자내에 존재하는 rs45431209 단일염기변이, 염색체 12번 내 MYH3 유전자내의 rs45431421 단일염기변이와 FLJ20920 유전자내의 rs45432261 단일염기변이, 그리고 염색체 13번에 RASA2 유전자내의 rs45431691 단일염기변이와 CXADR 유전자내의 rs45433249 단일염기변이 등 6개의 단일염기변이 마커가 발굴되었다(P<0.01). 세단계의 집단별 빈도 비교에서 동일하게 유의적인 빈도차이를 보인 KIAA0082 유전자는 사람에서 인터페론(interferon)를 증가(Up-regulation)시키는 유전인자로 연구되어 있으며 바이러스 침입의 저항에 연관된 유전자로 연구되어 있다[12-14]. 따라서 돼지의 KIAA0082 유전자 역시 추가적인 연구가 수행 되어져야 한다고 사료된다.
Table 5.
Annotation results of 22 SNPs associated with Mortality
| Reference ID |
SSC |
NCBI Assay ID |
Porcine gene name |
Position |
SNP code |
| rs45431209 |
7 |
ss23129507 |
KIAA0082
|
577,640 |
A/G |
| rs45431691 |
13 |
ss23130589 |
RASA2
|
67,128,049 |
A/C |
| rs45431421 |
12 |
ss23131519 |
MYH3
|
52,423,895 |
A/C |
| rs45435111 |
1 |
ss23132309 |
GSN
|
unknown |
A/G |
| rs45432261 |
12 |
ss23131659 |
FLJ20920
|
26,828,978 |
C/T |
| rs45433249 |
13 |
ss23131847 |
CXADR
|
131,088,764 |
A/G |
| rs10720198 |
16 |
ss16337513 |
DUSP1
|
48,837,371 |
A/G |
| rs45432566 |
6 |
ss23132764 |
WDR8
|
40,100,516 |
C/T |
| rs45432608 |
6 |
ss23132806 |
CNKSR1
|
77,206,489 |
C/T |
| rs45434874 |
15 |
ss23130072 |
NGEF
|
125,768,114 |
C/T |
| rs45435155 |
6 |
ss23130153 |
SESN2
|
61,933,658 |
C/T |
| rs45432669 |
6 |
ss23130767 |
CNOT1
|
19,267,073 |
A/T |
| rs45431071 |
10 |
ss23130469 |
EPRS
|
9,592,654 |
C/T |
| rs10720745 |
17 |
ss16338095 |
BMP2
|
191,959,283 |
A/T |
| rs45433249 |
13 |
ss23131847 |
CXADR
|
131,088,505 |
C/T |
| rs45431058 |
9 |
ss23130456 |
NCAPD3
|
unknown |
A/G |
| rs45435080 |
14 |
ss23132278 |
EWSR1
|
46,990,752 |
C/G |
| rs45431421 |
12 |
ss23131519 |
MYH3
|
52,423,983 |
C/G |
| rs45430738 |
7 |
ss23130336 |
DSP
|
4,931,933 |
A/G |
| rs10720341 |
13 |
ss16337664 |
RRP9
|
37,265,891 |
C/T |
| rs45431031 |
14 |
ss23130429 |
PES1
|
48,294,710 |
C/T |
| rs45432756 |
9 |
ss23131757 |
CTLA4
|
100,707,423 |
A/G |
Download Excel Table
위축돈과 정상돈 집단에서 유전자형 빈도 검증 결과
폐사추정돈과 정상돈 집단에서 유의적인 빈도차이를 나타낸 유전자내의 단일염기변이를 추가적인 검증연구를 위해 유의적인 효과가 있는 4개(DSP rs45430738G>A, RASA2 rs45431691A>C, MYH3 rs45431421C>A와 GSN rs45435111G>A)의 단일염기변이를 임의 선발하여 일반 삼원교잡 비육돈에서 태어난 자손들 중 위축돈 179두와 정상돈 200두 집단에 적용하였으며, 유전자형 분석은 PCR-RFLP method를 이용하여 실시하였으며 그 결과는 Table 6에 나타내었다.
Table 6.
Genotype and allele frequencies of DSP, RASA2, MYH3 and GSN genes polymorphisms in commercial pigs with susceptible and resistant allele
| Gene (“rs” number) |
Piglet population |
Genotype (N. of number) |
Total no. of pig |
Susceptible allele frequency |
Resistant allele frequency |
P-value |
| DSP (rs45430738) |
Susceptible |
AA: 17 (10%) |
AG: 54 (31%) |
GG: 104 (59%) |
175 |
A: 0.25 (0.58) |
A: 0.47 (0.52) |
<0.0001 |
| Resistant |
AA: 53 (27%) |
AG: 80 (40%) |
GG: 65 (33%) |
198 |
| RASA2 (rs45431691) |
Susceptible |
CC: 91 (51%) |
CA: 78 (44%) |
AA: 9 (5%) |
178 |
C: 0.73 (0.78) |
C: 0.83 (0.70) |
<0.0001 |
| Resistant |
CC: 145 (73%) |
CA: 42 (21%) |
AA: 12 (6%) |
199 |
| MYH3 (rs45431421) |
Susceptible |
AA: 45 (26.5%) |
AC: 80 (47%) |
CC: 45 (26.5%) |
170 |
A: 0.50 (0.79) |
A: 0.55 (0.72) |
NS |
| Resistant |
AA: 67 (34%) |
AC: 79 (41%) |
CC: 49 (25%) |
195 |
| GSN (rs45435111) |
Susceptible |
AA: 44 (25%) |
AG: 104 (59%) |
GG: 28 (16%) |
176 |
A: 0.56 (0.23) |
A: 0.33 (0.31) |
<0.0001 |
| Resistant |
AA: 29 (15%) |
AG: 72 (36%) |
GG: 98 (49%) |
199 |
Download Excel Table
상업돈의 위축돈 179두와 정상돈 200두를 이용하여 4개의 단일염기변이 마커를 검증하였는데 DSP rs45430738G>A, RASA2 rs45431691A>C와 GSN rs45435111G>A 세 개의 단일염기변이 마커는 위축돈 집단과 정상돈 집단에 유의적인 빈도차이를 나타내었다(P<0.001). 하지만 MYH3 rs45431421C>A 단일염기변이는 유의적인 빈도차이를 보이지 않았다(P>0.05). 그리고 DSP rs45430738G>A 단일 염기변이 마커는 폐사추정돈 집단과 정상돈 집단에서는 MA (minor allele)가 G 대립유전자로 나왔는데, 위축돈과 정상돈 검증집단에서는 MA가 A 대립유전자로 검출되었다. 그 이유로는 집단의 차이 또는 마커와 형질관의 연관 차이 등 여러 가지 요소가 반영될 수 있으므로, 향후 추가적인 연구가 필요한 것으로 사료된다.
DSP (dentin sialophosphoprotein)유전자는 사람에서는 상아질 이형성증에 깊게 관여하는 유전자로 치아의 발육장애를 일으키는 것으로 알려져 있으며, 돼지에서는 돼지 어금니의 기형장애를 유발하여 사료효율 및 식육장애를 일으켜 돼지의 성장발육에 치명적인 작용을 한다고 시사하였다[15, 16]. 또한 RASA2 (RAS p21 protein activator 2)유전자는 사람에서 모세혈관의 기형으로 인한 하지 비대의 원인이 되는 유전자로 림프기관의 기능 부전에 큰 영향을 미치는 것으로 알려져 있다[17, 18]. 그리고 GSN (gelsolin)유전자는 세포의 이동 및 번식능력에 영향을 주는 유전자로 사람에서는 암(cancer)세포에 관한 연구로 활발히 이루어지고 있다. 또한 아밀로이드증(amyloidosis)에 관여하여 영양장애, 신경세포장애 및 피부질환에도 영향을 미치는 유전자로 보고되어 있다[19, 20].
이렇듯 본 연구에서 선발된 유전자들은 질병 및 질환에 영향을 미치는 유전자들이 대부분이며 사람에서 많은 질병 연구가 되고 있다. 따라서 돼지에서도 이러한 유전자들이 추후 꼭 필요한 연구로 이루어져야 한다고 사료된다.
Acknowledgements
본 연구는 농림수산식품기술기획평가원의 생명산업기술개발사업 연구과제 "고효율 종돈계통조성 및 생산량 확대기술개발(과제번호: 311023-3)"의 지원으로 수행되었습니다.
REFERENCES
Albina E, Madec F, Cariolet R, Torrison J. Immune response and persistence of the porcine reproductive and respiratory syndrome virus in infected pigs and farm units. Vet Rec. 1994; 134 p. 567-573.

Alarcon P, Rushton J, Wieland B. Cost of post-weaning multi-systemic wasting syndrome and porcine circovirus type-2 subclinical infection in England - an economic disease model. Prev Vet Med. 2013; 110 p. 88-102.

Song D, Park B. Porcine epidemic diarrhoea virus: a comprehensive review of molecular epidemiology, diag-nosis, and vaccines. Virus Genes. 2012; 44 p. 167-175.

Charerntantanakul W. Porcine reproductive and respiratory syndrome virus vaccines: Immunogenicity, efficacy and safety aspects. World J Virol. 2012; 1 p. 23-30.

Mellencamp MA, Galina-Pantoja L, Gladney CD, Torremorell M. Improving pig health through genomics: a view from the industry. Dev Biol (Basel). 2008; 132 p. 35-41.

Lee YM, Alam M, Choi BH, Kim KS, Kim JJ. A whole genome association study to detect single nucleotide polymorphisms for blood components (Immunity) in a cross between Korean native pig and Yorkshire. Asian-Aust J Anim Sci. 2012; 12 p. 1674-1680.

Cho IC, Kang SY, Lee SS, Choi YL, Ko MS, Oh MY, Han SH. Molecular sexing using SRY and ZF genes in pig. J Anim Sci Technol (Kor). 2005; 47 p. 317-324.

Li XP, Hu ZL, Moon SJ, Do KT, Ha YK, Kim H, Byun MJ, Choi BH, Rothschild MF, Reecy JM, Kim KS. Development of an in silico coding gene SNP map in pigs. Anim Genet. 2008; 39 p. 446-450.

Li X, Kim SW, Do KT, Ha YK, Lee YM, Yoon SH, Kim HB, Kim JJ, Choi BH, Kim KS. Analyses of porcine public SNPs in coding-gene regions by re-sequencing and phenotypic association studies. Mol Biol Rep. 2011; 38 p. 3805-3820.

Tang K, Oeth P, Kammerer S, Denissenko MF, Ekblom J, Jurinke C, van den Boom D, Braun A, Cantor CR. Mining disease susceptibility genes through SNP analyses and expression profiling using MALDI-TOF mass spectrometry. J Proteome Res. 2004; 3 p. 218-227.

Baxter EM, Jarvis S, Palarea-Albaladejo J, Edwards SA. The weaker sex? The propensity for male-biased piglet mortality. PLoS One. 2012; 7 p. e30318.

Su AI, Pezacki JP, Wodicka L, Brideau AD, Supekova L, Thimme R, Wieland S, Bukh J, Purcell RH, Schultz PG, Chisari FV. Genomic analysis of the host response to hepatitis C virus infection. Proc Natl Acad Sci USA. 2002; 99 p. 15669-15674.

Geiss GK, Carter VS, He Y, Kwieciszewski BK, Holzman T, Korth MJ, Lazaro CA, Fausto N, Bumgarner RE, Katze MG. Gene expression profiling of the cellular transcriptional network regulated by alpha/beta interferon and its partial attenuation by the hepatitis C virus nonstructural 5A protein. J Virol. 2003; 77 p. 6367-6375.

Guerra S, López-Fernández LA, Pascual-Montano A, Muñoz M, Harshman K, Esteban M. Cellular gene expression survey of vaccinia virus infection of human HeLa cells. J Virol. 2003; 77 p. 6493-6506.

Yamakoshi Y, Hu JCC, Fukae M, Iwata T, Kim JW, Zhang H, Simmer JP. Porcine dentin sialoprotein is a proteoglycan with glycosaminoglycan chains containing chondroitin 6-sulfate. J Biol Chem. 2005; 280 p. 1552-1560.

Yamakoshi Y, Hu JC, Fukae M, Iwata T, Kim JW, Zhang H, Simmer JP. Porcine dentin sialoprotein is a proteoglycan with glycosaminoglycan chains containing chondroitin 6-sulfate. J Biol Chem. 2005; 280 p. 1552-1560.

Hershkovitz D, Bergman R, Sprecher E. A novel mutation in RASA1 causes capillary malformation and limb enlargement. Arch Dermatol Res. 2008; 300 p. 385-388.

Boon LM, Mulliken JB, Vikkula M. RASA1: variable phenotype with capillary and arteriovenous malformations. Curr Opin Genet Dev. 2005; 15 p. 265-269.

Chastan N, Baert-Desurmont S, Saugier-Veber P, Dérumeaux G, Cabot A, Frébourg T, Hannequin D. Cardiac conduction alterations in a French family with amyloidosis of the Finnish type with the p.Asp187Tyr mutation in the GSN gene. Muscle Nerve. 2006; 33 p. 113-119.

Tsai MH, Wu CC, Peng PH, Liang Y, Hsiao YC, Chien KY, Chen JT, Lin SJ, Tang RP, Hsieh LL, Yu JS. Identification of secretory gelsolin as a plasma biomarker associated with distant organ metastasis of colorectal cancer. J Mol Med (Berl). 2012; 90 p. 187-200.