Introduction
18세기 초반 나타난 1차 산업혁명 이후 4번째로 출현한 4차 산업혁명은 정보통신기술의 융합으로 이루어지는 차세대 산업혁명으로 정의되는데, 물리적, 생물학적, 그리고 디지털적 세계를 인공지능(artificial intelligence)에 입각해서 통합시키고, 산업의 여러 분야에 영향을 미치는 다양한 신기술로 정의될 수 있다. 4차 산업혁명 기술의 눈부신 발달로 인해 의학과 산업영역에서 인공지능의 사용빈도가 비약적으로 성장하고 있다. 이 인공지능 기술은 보통 머신러닝, 딥러닝, 자연어 처리와 이미지 인식기술 등으로 구분되고 있다[1].
역사적으로 인공지능의 발달이 잘 이루어지지 않았던 시대에는 충분한 컴퓨터 능력이 없어서 전반 분야의 자료를 디지털화하기가 어려웠으나, 컴퓨터의 처리능력이 비약적으로 발달함에 따라 디지털화가 가능하게 되었다. 이 인공지능 기술은 반복적이고 세부적인 작업을 빠르고 정확하게 완료하는데 있어서 인간보다 우수하다는 것이 증명되었고, 일관성(consistency), 정확성(accuracy)과 확장성(scalability)을 기반으로 점차 다양하고 복잡한 영상 분석 및 진단 보조 솔루션으로 그 활용성이 확산되고 있다. 특히 독성병리 분야에서 화학물질, 의약품, 의료기기, 화장품과 농약 등의 평가에서 있어서 인공지능을 이용한 정교한 평가가 가능할 것으로 기대되고 있다[2].
인공지능의 개념
인공지능은 컴퓨터를 이용하여 사고, 학습과 자기 개발 등을 할 수 있는 방법을 연구하는 컴퓨터 공학의 한 분야로 컴퓨터가 지능적인 행동을 모방할 수 있도록 하는 것을 지칭한다. 이 인공지능은 그 컴퓨터공학 분야로만 존재하는 것이 아니라, 다른 여러 분야와 관련을 맺고 있어서 이런 기술을 도입하여 해당 분야의 문제를 해결하려는 노력이 활발하게 진행되고 있다.
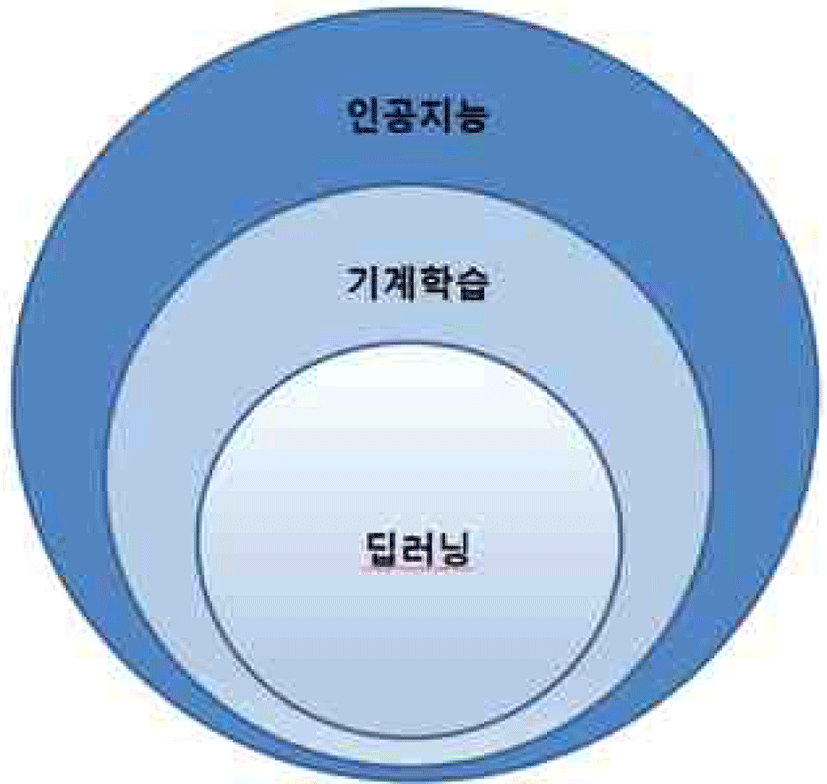
인공지능은 그 영역상에서 머신러닝이라는 세부 영역을 가지며, 머신러닝 안에 딥러닝이라는 세부 영역을 갖는다(Fig. 1).
인공지능 학습은 지도학습, 비지도학습과 강화학습으로 구분할 수 있다. 지도학습에서는 컴퓨터에게 대상의 레이블(예: 개체명, 동물종, 연령과 병명 등)을 제공하여 정답을 최대한 맞추도록 알고리즘을 훈련하는 과정을 말한다. 지도학습에는 세 가지 데이터 세트가 사용되는데, 트레이닝 세트(training set), 검증 세트(validation set)와 테스트 세트(test set)가 있다. 트레이닝 세트를 이용하여 컴퓨터가 코딩된 알고리즘을 이용하여 최대한 정확도를 높일 수 있도록 훈련시킨다. 예를 들어, 콩팥을 식별하기 위해 토리, 콩팥 세관과 같은 정상 구조를 라벨하여 컴퓨터가 알고리즘을 통해 학습할 수 있도록 하며, 이 학습된 알고리즘을 수정 보완하여 콩팥의 구조를 최대한 정확하게 식별하도록 훈련을 반복하는 것이다. 이 훈련된 알고리즘의 성능은 검증 세트를 사용하여 학습 정확도가 어느 정도 되는지 확인하게 된다. 이 때 검증 세트는 트레이닝 세트와 구별되며, 레이블이 지정된 데이터를 포함한다. 마지막으로, 학습된 알고리즘이 작동하는지 테스트 세트에서 독립적 작업을 수행하여 사용한 알고리즘의 정확도를 산출한다. 예를 들면, 라벨이 없는 이미지를 테스트 세트에 사용하고, 그 테스트 세트가 학습된 알고리즘을 통해 콩팥 구조를 정확하게 분류하는가를 확인하는 것이다.
비지도학습의 경우는 생물학적 조직의 특징을 정의하는 것이 어렵거나 불가능한 경우에 사용된다. 이 접근 방식에서 컴퓨터는 다양한 장기의 다양한 조직 자료가 함께 제공되며, 컴퓨터는 조직의 패턴을 식별하고, 유사한 패턴을 가진 패턴을 함께 클러스터링한다. 비지도학습의 전반적인 목표는 출력에 대한 사전 사용자 정의 없이 입력 데이터의 구조를 식별하여 데이터 기반 및 가설 생성 접근 방식으로 만드는 방법이다[3]. 비지도 학습에서 식별된 패턴은 답을 제공할 뿐만 아니라, 조사자가 생각하지 못한 질문을 제기할 수 있다. 비지도 학습의 일반적인 적용은 유전학, 전사체학, 단백체학과 대사체학 등의 질병 상태 간의 복잡한 상호 관계를 탐색하는 것이다.
강화학습은 컴퓨터가 스스로 실수를 통해 학습을 진행하는 방식을 말한다. 이 접근 방식에서는 컴퓨터에 레이블이 지정되지 않은 입력이 제공되고, 적대적 신경망을 통해 출력을 예측해야 하고, 예측이 잘못된 경우 컴퓨터에 올바른 레이블이 제공되며, 컴퓨터는 이러한 데이터를 향후 예측에 사용한다[4]. 또한 강화학습은 입출력 쌍으로 이루어진 트레이닝 집합이 제시되지 않으며, 잘못된 행동에 대해서도 명시적으로 정정이 일어나지 않는다는 점에서 일반적인 지도학습과 구별된다.
기계학습은 컴퓨터가 명시적인 프로그래밍 없이 스스로 학습하고 경험을 통해 지속적으로 학습하여 성능을 향상시킬 수 있는 능력을 제공하는 인공지능내의 분야이다[5]. 딥러닝은 기계학습의 세부영역이며, 신경망을 통해 지도 혹은 비지도학습 패턴으로 학습하는 것을 말한다. 딥러닝에서는 입력층을 통해 데이터 정보를 입력하고, 출력층을 통해 최종 결과를 출력하는데, 이 입력층과 출력층 사이에 많은 ‘숨겨진 층들(hidden layers)’이 존재한다. 이 변환은 정보가 다음 레이어로 이동하는지 여부와 방법에 영향을 주는 한 레이어에 할당된 가중치와 편향을 통해 발생하며, 출력 계층에는 계산된 출력이 포함되도록 한다. 이런 구조를 통해 컴퓨터는 데이터 세트를 검토하여 학습 방법을 스스로 조정할 수 있다.
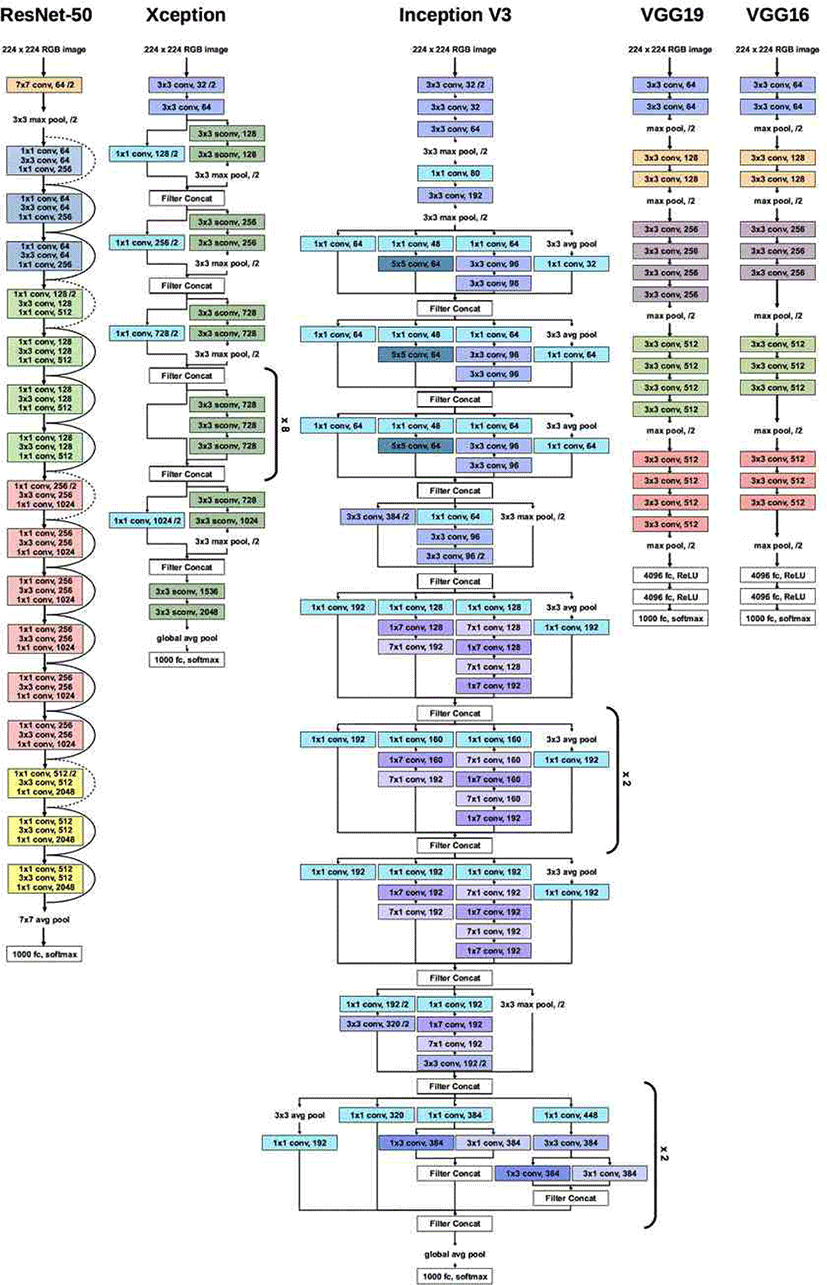
최근 몇 년 동안 딥러닝 기술을 활용하여 컴퓨터 비전 연구가 빠르게 발전하면서 현미경 영상을 자동화된 패션별로 분류하고 계산하는 기술이 적용되기 시작했다[6]. 딥러닝의 진보가 이미지 분류와 구분에서 높은 성과를 거두면서 의료 이미지 분석 분야로 영역이 확대되었는데, 여러 딥러닝 기술 중에서 합성곱 신경망(convolutional neural network, CNN)은 사실상 이미지 인식의 표준이 되었으며, 여러 작업에서 인간의 성능에 접근하고 있다[7]. CNN은 종종 수백만 개의 이미지를 포함하는 거대한 이미지 트레이닝 세트에서 직접 관련 요소를 학습하여 작동하며, 다양한 알고리즘이 개발되었고, 여러 단계를 거쳐 학습이 진행된다(Fig. 2).
대표적인 CNN인 인셉션 구조는 다중 스케일과 다른 특징을 가진 피처 맵을 동시에 처리할 수 있다는 장점이 있으며, 분류를 위한 효율적인 도구이다[8]. 딥러닝 모델인 Inception v3를 이용하여 97% 정확도로 폐암을 분류하였고, 신경망 모델이 돌연변이 프로필과 결합된 폐샘암종 환자의 영상에 대해 훈련되었을 때 특정 유전자의 돌연변이가 존재한다고 예측이 가능하였다[9]. 이후 VGGNet, ResNet과 Alexnet 등을 사용하는 CNN을 기반으로 하는 다른 합성 신경망이 제안되었고, 이에 대한 성능이 테스트되었다[10].
특히, 딥러닝은 클래스 분리성을 최대한 확보하기 위해 기본 데이터의 학습된 표현을 반복적으로 개선하여 결과 값을 조정한다. 딥러닝을 통해 입력 데이터에서 나타나는 패턴 특징을 학습하고, 이를 반복적으로 객체의 특징을 구별하여 분류하는 작업이 진행되며, 데이터 세트의 분류 결정을 반복적으로 수행한다. 이후 검증 세트와 테스트 세트로 학습을 진행하여 학습된 알고리즘의 정확도를 산출하게 된다. 독성병리 영역은 시각적 이미지를 분류하는 분야가 많으므로 CNN은 이미지의 해석 및 시각화에 매우 유용하다고 볼 수 있다.
전통적 독성병리와 인공지능 기술 도입 배경
독성병리 기술은 새로운 물질의 효능(efficacy)과 안전성(safety)을 평가하는데 직접적으로 가장 정확하게 병변을 판독하는 분야이며, 질병의 진단 및 원인 규명에 중요한 역할을 하고, 치료법 개발에 합리적인 근거와 방향을 제시하는 역할을 하고 있다.
일반적으로 독성병리 진단을 위해서는 조직의 채취, 고정, 절취, 조직처리, 포매, 박절, 염색, 봉입, 현미경 검경과 진단의 과정을 거쳐야 한다. 조직의 고정에는 시험하는 목적에 따라 고정제를 선택하여 사용하여야 하며, 일반적으로 10% 중성포르말린을 사용한다. 이후 조직을 일정한 크기로 잘라주는 절취 과정을 거치고, 조직카세트에 넣은 후, 탈수, 투명 및 침투의 조직처리과정을 거치고 포매를 통해 조직 블록을 제작한다. 조직 블록은 미세박절기를 사용하여 박절하며, 박절된 표본은 염색 과정과 봉입을 거쳐 조직슬라이드로 제작되며, 이 조직슬라이드를 현미경으로 검경하여 병변을 진단한다.
이런 조직검사의 가장 큰 목적은 병변의 정확한 진단이다. 독성병리 연구자들은 병리학적 병변을 찾기 위해 현미경으로 조직 슬라이드를 조사하여야 하며, 검사 중에 많은 조직슬라이드를 검경하여 병변을 찾아야 하는 부담을 안고 있다. 독성병리 연구자가 연구당 약 100–10,000장의 조직슬라이드(약 300–300,000개의 조직)를 현미경으로 검토하여야 하는데, 이는 노동 집약적인 작업이며, 조직 슬라이드를 검경하는데 시간이 많이 걸린다. 특히 발암성시험은 안전성 평가에 사용되는 가장 복잡한 시험지침 중 하나이며, 독성병리 연구자는 하나의 발암성시험에서 약 10,000장 이상의 조직슬라이드(약 30,000개 이상의 조직)를 검사해야 한다[11].
진단을 위한 병리 슬라이드의 양이 폭발적으로 증가하고 있지만, 현재 비임상 또는 임상분야에 종사하는 연구자를 위한 교육시스템이 이런 사회적 요구를 모두 충족시키지 못하고 있다[12]. 또, 정확한 진단을 위해서는 독성병리연구자가 관련 진단명을 학습하고, 적지 않은 시간을 투자하여 진단과 관련된 일을 수행하고, 숙련된 경험을 바탕으로 독성병리전문가 자격증을 획득하여야 하며, 병변을 판독하는데 개인 편차를 줄이기 위해서는 보다 체계적인 교육시스템이 필요하다.
독성병리 분야에 인공지능의 도입 효과
독성병리 분야에 있어서 인공지능기술을 활용하면 아래와 같은 효과가 나타날 것으로 기대된다.
첫째, 인공지능기술을 사용하여 조직슬라이드에서 비정상 샘플을 분류하는 선별 작업을 수행하여 독성병리연구자가 현미경으로 병변을 스크리닝하는 시간을 줄일 수 있다. 인공지능을 사용하게 되면 독성병리연구자가 모든 조직슬라이드를 검경하지 않아도 되고, 이에 따른 병변 판단에 조직슬라이드 판독에 필요한 시간과 경비가 많이 줄어들 것으로 기대된다[13]. 또, 조직 슬라이드를 보다 정밀하게 진단할 수 있는 시간적 여유를 확보하여 이에 따라 진단 정확도가 높아질 수 있을 것이다.
둘째, 인공지능기술을 사용하여 진단 정확도를 높일 수 있다. 독성병리연구자 개인간의 진단에 대한 편차가 존재하며, 이런 편차에 의해 진단불일치가 발생할 수 있는데, 인공지능을 사용하면 독성병리연구자가 능력이 질적으로 향상되고, 진단의 정확성과 일관성이 높아질 것으로 기대되고 있다[14]. 특히 인공지능을 사용에 의해 병변의 등급이 객관적으로 부여될 수 있다면, 정량적 등급 시스템을 일괄적으로 적용할 수 있고, 독성병리연구자들의 차이 및 실험실 간의 차이를 줄이는데 도움이 될 수 있을 것으로 기대된다.
셋째, 독성병리연구자가 인공지능 분석을 통해 시험물질에 대한 독성기전을 파악하고, 유사물질에 대한 자료에서 발생한 병변 등의 사례를 빠르게 파악하여 검경에 활용한다면 시험물질에 대한 독성병리보고서와 독성보고서 등의 품질이 크게 향상될 것으로 기대된다.
독성병리자료의 디지털화 필요성
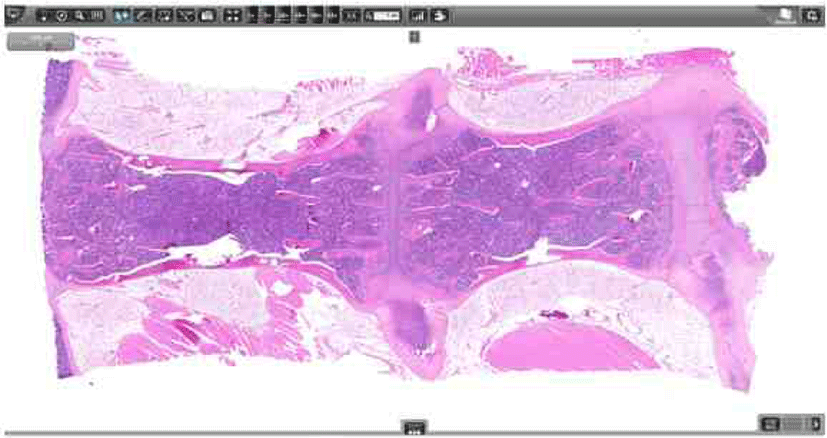
독성병리 자료의 대표적인 것이 유리 슬라이드 위에 놓인 조직을 올려 놓은 조직슬라이드인데, 아날로그 형태인 조직을 슬라이드 스캐너(virtual slide scanner)를 사용하면 디지털 형태의 이미지로 바꿀 수 있으며, 이것이 전체 슬라이드 이미지(whole slide image, WSI)이다(Fig. 3).
이 WSI 기술은 조직 슬라이드를 디지털화하여 병리학 평가를 수행할 수 있는 수단으로 자리잡고 있으며[15], 병리 검경, 연구 및 교육 분야에서 WSI를 사용한 가상 현미경 채택으로 전환되고 있다[16]. 또, WSI를 이용한 이미지 처리 기술을 통해 분석을 자동화하고 최적화하기 위한 노력이 진행되어 왔다[17]. 또한, WSI 기반 기술을 이용하여 독성병리 연구자의 잠재적인 편견을 없애고, 시간 효율성을 높이고 비용을 줄이기 위한 방법들이 다양하게 시행되고 있다[18].
슬라이드 스캐너는 광학현미경, 카메라, 저장시스템과 컴퓨터 하드웨어 등으로 구성되어 있는데, 스캐닝을 수행하는 시간은 유리슬라이드 1.5 mm2 면적과 20× 배율 기준으로 할 때 슬라이드에 놓인 조직 크기에 따라 다르나, 대개 슬라이드 한 장당 1–5분 정도 소요된다. 독성병리에 사용되는 슬라이드 스캐너는 3D 히스텍사(파노라믹), 라이카(아페리오)와 로슈(벤타나) 등의 제품이 주로 사용되고 있다. WSI를 저장할 때의 파일 포맷이 슬라이드 스캐너 공급업체마다 다르기 때문에 각 회사 별로 WSI뷰어 프로그램은 각각의 파일 포맷을 지원하는 형태로 스캐너와 함께 제공되고 있다. 현재 슬라이드 이미지 뷰어는 독성병리연구자가 현미경을 볼 때처럼 가상 슬라이드를 이미지를 디스플레이 상에서 보여줌으로써 이미지의 확대, 축소와 주석 달기가 가능하고, 길이와 크기의 형태학적 측정 등이 가능하도록 구성되어 있다. 또한, 원격 회의를 할 수 있도록 설계되어 있어 의료계에서는 원격 진단에 활용되고 있다. 슬라이드 스캐너와 연동해서 WSI를 분석하는 소프트웨어를 판매하는 곳은 3D 히스텍사와 라이카 등이 있고, WSI 이미지를 분석하는 소프트웨어만을 판매하는 회사로는 데피니언스(Definiens), 비지오팜(Visiopharm)과 인디카랩(Idica Lab.) 등이 있다.
디지털화된 조직슬라이드에서 생성된 정보를 기반으로 데이터 관리에 중점을 두어 연구하는 분야가 디지털병리(digital pathology)이며, 이에는 디지털 이미지 관찰, 분석과 저장 등을 하며, 데이터에서 생물학적 및 임상적으로 관련된 정보를 추출하고, 수학적 모델을 사용하여 진단 추론 및 예측을 생성할 수 있다. 디지털병리에는 현미경이 아닌 컴퓨터 모니터 등의 영상표시장치를 통해 나타내는 WSI, 네트워크를 이용해 다른 장소에 위치한 전문가와 디지털 영상을 공유하거나 소견을 구하는 원격병리(telepathology) 그리고 영상분석 소프트웨어를 이용한 정량분석 등이 포함된다.
이것에는 잠재적으로 비임상 및 임상 진단, 다양한 화학물질 평가 및 신약 개발 등에서 병리학 기반 시험의 입력 기준을 통일하고, 시험 종말점에 대한 정확도를 높이며, 실험의 재현성을 확보하고 독성병리 진단표준화를 높일 것으로 기대된다[19]. 또한, 현재 인공지능을 적절하게 사용한다면 의료 영상, 특히 디지털 병리 분야에서 획기적인 잠재력을 나타낼 것으로 판단된다[20]. 이와 관련하여 여러 기업들이 조직의 병변에 대한 디지털 슬라이드를 스크리닝하는 인공지능 알고리즘을 개발하고 있다.
이런 알고리즘은 특히 대규모의 조직 샘플을 이용하여 추가 검증이 필요하지만, 향후 비임상독성시험에서 일반적으로 관찰되는 특정 병변의 분류를 자동화하는데 사용할 수 있다. 독성병리학 분야에서 인공지능 알고리즘은 이미 큰 가능성을 보여주기 시작했으며, 알고리즘의 수가 증가한다는 것은 인공지능을 이용하여 독성병리 플랫폼이 정밀해지고, 개인별 맞춤 의학에 있어서 진단의 정확성을 더 높일 수 있다는 것을 의미한다[2].
독성병리 연구에서 인공지능 활용 분야
인공지능의 발달로 독성병리 연구에 있어서 병변의 분석을 보다 효과적으로 달성할 수 있으며[21], 병리 분야에 인공지능을 접목한 다양한 방법들이 개발되고 있다[22].
독성병리분야에서는 여러 인공지능 분류기를 사용하여 병변을 분류하고자 하는 노력을 하고 있으며, 인공지능을 활용하여 정확하고 객관적인 병변의 분류 및 등급 지정이 가능하게 되었다. 또, 병변을 판독하기 위해 병리전문가가 인공지능과 협업을 한 경우가 병리전문가 단독 혹은 인공지능 단독으로 진단한 경우의 정확도보다 더 높은 것으로 나타났다. 예를 들면, 암 검출에서 인공지능 알고리즘을 사용한 경우, 병리 전문가와 동등하거나 약간 더 나은 결과를 도출하였다고 보고되었고[23], 인공지능 알고리즘을 사용하여 병리 전문가와 유사한 정확도로 사람 피부암 생검의 조직슬라이드에 대한 진단이 가능하게 되었다[24].
검출한 병변을 시각화하여 병변 진단을 원활하게 진행할 수 있는데, 인공지능을 사용하여 유방종양의 양성과 악성을 구별할 수 있다고 보고되었다[25]. 또한, 인공지능을 사용하여 핵 분할, 상피 분할과 관 구조 분할 등과 같은 여러 기능을 수행하며, 연구자가 놓칠 수 있는 여러 병리학적 파라미터의 상호 관계를 알아낼 수 있다고 보고되었다[10].
독성병리 분야와 관련된 WSI를 충분히 확보하고, 인공지능을 이용하여 병변과 유사한 이미지를 손쉽게 검색하게 된다면 슬라이드 검경시간이 단축되고, 정확한 병리진단이 가능할 것으로 예상된다. 예를 들면, 까다로운 종양 진단의 경우 감별 진단을 위해 오랜 시간이 소요될 수 있으나, 웹상의 WSI 저장소를 통해 종양 소견들을 축적해 두고, 추후에 비슷한 사례에 대해 검색이 가능하다면 감별진단을 위한 고민을 많이 해소할 수 있을 것이다.
독성병리학적으로 병변을 진단하는 경우, 병변 정도를 상대적으로 등급화하여 평가하며, 일반적으로 다음과 같은 6가지 단계로 구분하는데, 이에는 병변 없음(no lesion, 0점), 매우 미약한(minimal, 1점), 미약한(mild, 2점), 중간(moderate, 3점), 심한(severe, 4점), 매우 심한(massive, 5점)이 있다. 효능과 안전성시험에서 이 6단계의 평가는 대략적인 정성분석은 가능하지만, 정량분석에는 어려움이 종종 나타난다. 특히 신약에 대한 효능을 분석하는데 있어서 정성분석을 도입할 경우, 치료제 투여군이 대조군과의 차이가 크게 나지 않아 어려움을 겪는 일이 많이 발생하고 있다. 이런 경우, 인공지능을 통한 정량분석을 적용하면 평가에 대한 지표 수치의 세분화가 가능하고 효능 및 안전성을 효과적으로 평가할 수 있다는 장점을 가질 것으로 기대된다.
먼저, 인공지능을 이용하여 조직내에 있는 세포를 분리하고 정량화하여 이를 수치화할 수 있다. 예를 들면, 골수에 대한 독성을 평가하는데 있어서 골수 세포를 자동으로 계수하여 골수의 저하 등을 판독하는데 효율적이었다고 보고되었다[26]. 또, 랫드의 골수에서 자동 이미지 분석 방법을 사용하여 골수 세포밀도를 정량화하였고, 이 자동화된 방법은 골수 고갈을 수동으로 채점하는 것보다 빠르고 민감한 것으로 보고되었다[27]. 또한, 인공지능 알고리즘은 유사분열상을 정량할 수 있으며[28], 형태학적 기반으로 혈구세포를 분류할 수 있다고 보고되었다[29].
인공지능을 이용하여 조직내에 있는 구조물을 구별하여 세절화하여 이를 수치화할 수 있다. 예를 들면, 콩팥의 토리를 자동으로 계수하기 위하여 인공지능 기반 처리방법을 개발하고, 이후 토리의 수를 측정하는 정량 분석이 가능하게 되었다[30]. 또, CNN 기법으로 만성 신장 질환 환자로부터 마손삼색(Masson trichrome) 염색된 콩팥 조직에서 토리를 정밀하게 검출하고 분할할 수 있다고 보고되었다[31]. 또한, 인공지능분석을 통해 콜라겐을 정량화할 수 있으며, 특정 조직 유형을 추정할 수 있다고 보고되었다[32].
인공지능기법은 특수염색과 면역조직화학염색 결과의 정량적 해석에 효율적이다[33]. 인공지능기법은 면역염색 슬라이드를 정량화하는데 사용된다[34]. 예를 들면, CNN 알고리즘을 사용하여 흑질에서 도파민 뉴런의 계수화가 가능하였다고 보고되었다[35]. 또, CNN을 사용하여 마우스 폐 섬유화 모델에서 섬유화 정도를 점수화하였고[36], 폐 디지털 이미지에서 폐 조직 밀도를 기반으로 섬유화의 정량화가 가능하게 되었다고 보고되었다[37]. 또한, WSI 기반 이미지 분석 도구를 통해 사용자는 기술을 활용하여 이전에는 사람이 수동으로 수행하기 번거롭거나 심지어 불가능한 작업을 수행할 수 있게 되었는데[38], 이에는 간섬유화[39], 신장섬유화[40], 폐섬유화[41], 심장섬유화[42]와 췌장 β세포 mass[43] 등의 세포학적 정량 평가가 있다.
독성병리 진단방법은 오래동안 사용되어 왔고, 바이오 분야에서 보편화된 방법이지만 단편적인 2차원 조직 정보만을 제공하고, 넓은 부위의 조직정보들을 획득하는 데는 한계가 있다. 특히 병변의 유무를 판단함에 있어서 시험물질이 체내에 투여된 경우 분포가 전체적으로 균등하게 이루어지며, 병변이 장기의 표면 혹은 내부에 균등하게 분포하고 있으며, 병리학적으로 조직 단면을 관찰할 때 전체를 대표한다고 가정하고 검사를 한다. 하지만, 최근에 시험물질의 종류가 다양해지고 있고, 시험물질에 따라 분포 자체가 균일하다는 전제를 할 수 없는 경우도 나타나고 있다. 예를 들면 동물모델을 사용한 항암제 평가에 있어서 침윤성 종양을 조사할 경우, 단면에 따라 종양의 면적이 달라질 수도 있고, 경우에 따라서는 종양 단면이 검출되지 않을 가능성이 있기 때문에 2차원 절편에서 효능 평가를 하는 것보다는 3차원 평가로 진행하는 것이 합리적이라고 생각된다.
최근에 바이오이미징 기술이 발달하면서 조직의 구조를 2차원뿐만 아니라, 3차원으로 구현할 수 있는 것이 가능해지고 있고, 최근 과학 기술의 발달로 인해 해상도가 점점 증가되어 광학현미경에 근접하여 입체구조를 증명할 수 있다고 보고되었다[44]. 또, 바이오이미징, 독성병리와 인공지능 기술을 결합할 경우 3차원 상태에서 병변의 위치를 파악할 수 있으며, 볼륨이미징(volumetric imaging)을 통해 입체적인 구현이 가능하며, 특수 세포를 조직 내에서 추적할 수 있다는 장점을 갖는다[45]. 최근 병변을 절단할 때 인공지능 기술을 이용하여 각 섹션을 스캔하는 2차원의 이미지를 통해 3차원의 이미지를 구성하는 것이 가능하게 되었다[46]. 또한, 3차원 기법을 활용하여 C57BL/6 마우스 뒷다리의 미세 혈관 구조를 특성화하고[47], 자궁 경부 암종의 침습을 평가하는 것이 가능하게 되었다[48].
인공지능 기법을 사용하여 일반염색 슬라이드 이미지를 조사하고, 다른 복잡한 유전체, 전사체 및 대사체 결과를 통합하면 연구자가 분자 레벨에서의 분석이 가능하며, 이와 같은 자료를 혈액 및 소변과 같은 임상 데이터와 결합하여 환자의 생존율 등의 질병 예측이 가능할 것으로 생각된다. 예를 들면, 대장암 조직 분석을 통해 환자의 생존율을 예측할 수 있었으며[49], 인공지능을 사용하여 중피종 병변이 주로 기질에 위치해 있고, 염증, 세포 다양성, 공포화와 관련된 조직학적 특징을 분류하여 환자 생존율을 예측할 수 있다고 보고되었다[50]. 또, 인공지능으로 암조직내의 조직분석을 통해 에스트로겐 수용체와 환자의 생존율의 상관관계를 규명하였고[51], 폐암 조직분석을 통해 핵의 양상과 환자의 생존율과의 상관관계를 밝혔다[52].
독성병리학적 이미지와 유전자 프로파일을 결합하여 분석함으로써 유전자 돌연변이와 반복서열불안정성(microsatellite instability, MSI) 예측이 가능하다. 예를 들어 인공지능을 이용하여 분석한 결과, 임상 결과 및 돌연변이 유전자를 예측할 수 있다고 보고되었다[53]. 또, 인공지능 알고리즘을 이용하여 폐암 아형을 탐지하고 분류하는데 정확할 뿐만 아니라, 병리학 이미지를 기반으로 암 특이적 유전자 돌연변이를 예측할 수 있다고 보고되었다[9]. 또한, 인공지능 기법을 사용하여 위장관암의 조직검사 슬라이드 분석자료와 임상결과를 통합적으로 분석하여 MSI를 예측할 수 있다고 보고되었다[54].
이러한 인공지능 알고리즘을 사용할 경우, 조직 슬라이드 자체에서 분석이 가능하여 유전체 분석 등의 샘플 처리 시간을 단축하고, 병리전문가의 작업량을 줄이고, 진단 검사당 비용을 감소시키면서 진단 감도를 높일 수 있는 잠재력을 가지고 있다고 볼 수 있다.
독성병리 피어리뷰는 병변 진단의 정확성과 일관성, 그리고 결과의 객관적 평가를 내리기 위하여 1차 병리전문가가 검사한 병리자료를 경험이 풍부한 다른 병리전문가가 점검하여 보완하는 과정이다[55]. 피어리뷰를 진행하기 위해서는 조직 및 병변의 확인, 조직 및 병변의 검사, 진단의 불일치 해결, 병리진단자료의 수정 및 교정, 그리고 병리보고서의 검토를 진행하고, 최종적으로 피어리뷰 보고서를 작성하여야 한다. 피어리뷰의 결과, 1차로 검경한 병리전문가와 피어리뷰 병리전문가 사이에 이견이 해결되지 않을 경우에는 병리전문가 협의체(Pathologist Working Group, PWG)를 소집하여 진단의 이견을 해결한다.
현재 병리 피어리뷰는 국내 몇 개 시험기관에서만 실시하고 있는 것이 현실인데, 병리보고서의 국제적 위상을 확보하기 위해서는 국내에 있는 모든 시험기관에서 생성된 병리보고서에 대한 독성병리 피어리뷰를 도입하여 정착시키고 확대하여 나가는 것이 중요하다. 독성병리전문가에 의한 병변 진단에 오류가 발생하였거나, 독성병리전문가간의 진단 의견이 상충되는 경우, 인공지능을 통해 피어리뷰를 진행할 경우, 독성병리 피어리뷰 프로세스에 필요한 시간을 상당히 줄일 수 있을 것으로 기대된다.
많은 화학물질의 출현과 다양한 생물학적 제재의 개발 등에 의해서 독성시험을 수행하는 국내외 여러 기관에서 독성병리 판독에 대한 수요가 늘어나고 있어서 독성병리전문가의 양성에 대한 요구가 최근 증가되고 있다. 현재 국내에는 훈련된 독성병리전문가가 부족하고, 진단명의 다양성으로 인한 진단 오류가 생기고 있고, 판독자 사이에, 그리고 판독자 자신에게 편차가 존재하고 있다. 이러한 제반의 문제점을 개선하기 위하여 기존의 전통적인 병리업무와 인공지능 기술을 접목하여 개발한 새로운 독성병리 교육시스템이 필요하다는 요구가 대두되고 있는 실정이다.
기존 조직슬라이드는 보관의 장소적 한계와 염색의 퇴색 등에 의한 자원의 소실 위험이 있는데 비해, WSI는 자료의 영구적 보관이 가능하고, 픽셀에 저장된 영상 값을 통해 여러 정보를 분석할 수 있는 장점을 갖는다. 이런 장점을 충분히 살려서 다양한 병변을 포함한 독성병리 슬라이드를 WSI 형태로 최대한 많이 구축하고, 인공지능을 이용하여 독성병리 교육시스템을 체계적으로 구축한다면 이 교육시스템을 통해 많은 병변을 접하게 되고, 실력 있는 독성병리전문가의 빠른 양성이 가능해질 것으로 생각된다. 또한, 온라인 교육에서 WSI 자료를 활용할 경우, 시간, 공간과 교육 인원의 참여 수에 제한이 없고, 공간적인 제약을 받지 않고 교육을 진행할 수 있다는 장점을 갖는다. 2016년 인공지능 알파고가 이창호 9단을 이겼을 때 바둑 기원의 반응이 두 가지였다고 한다. 이제 바둑은 인공지능이 정복했으니 인간은 경쟁이 안되니 바둑 시장은 없어진다고 보는 시각이 있었고, 한편에서는 오히려 바둑을 배우는 기사가 인공지능을 통해 학습하게 되니 바둑 실력이 전체적으로 늘어난다고 했다. 판세는 후자로 되어 가고 있는 양상이다. 바둑을 배우는 사람이 증가하고, 바둑을 배울 때 인공지능을 활용하여 실력을 더 빨리 쌓아가는 시대로 가고 있는 것이다. 독성병리 교육용 자료를 체계적으로 저장하여 인공지능을 통해 필요로 할 때 사용할 수 있는 시스템을 만든다면 새로운 기술을 접목하여 더 유능한 독성병리전문가를 양성할 수 있는 시대가 열릴 수 있을 것으로 생각된다.
독성병리 연구 수행에 있어서 조직 슬라이드 자료와 함께 체중, 장기 무게, 혈액학적 결과, 혈액생화학적 결과와 뇨 검사 결과 등의 많은 시험 데이터가 생성된다. 이와 같은 시험 데이터를 동물별로, 그리고 장기별로 일괄적으로 통합하여 빅데이터를 구축하는 것이 중요하다. 빅데이터는 기존 데이터베이스 관리 도구의 능력을 넘어서는 대량의 정형 또는 비정형의 데이터 집합도 포함하는 데이터로부터 가치를 추출하고 결과를 분석하는 기술이다. 독성병리분야에서 빅데이터가 구축되면 이것을 이용하여 발견된 병변과 관련된 유사 이미지를 빠르게 검색할 수 있으며, 다른 임상병리 자료와 함께 결합하여 분석하면 시험물질에 대한 효능 및 안전성에 대한 충실한 해석이 가능할 것으로 생각된다. 특히, 진단영역에서는 현미경을 대체하는 수단으로서 WSI에 대한 기관의 규제를 충족하기 위한 노력들이 지속되고 있고, 가까운 미래에 WSI를 통한 진단이 가능할 것으로 보고 있는데, WSI는 그 자체로 큰 디지털 데이터이기 때문에 WSI 이미지로부터 의미 있는 정보를 추출하기 위해서는 빅데이터 분석기법과 인공지능기술을 필요로 한다.
독성병리학 분야에서 생성된 WSI 등의 시험자료를 빅데이터 형태로 통합하여 구축하고, 인공지능을 폭넓게 사용하려면 공개적으로 사용 가능한 WSI를 확보하고, 라벨링 분류를 통해 정확도를 향상시켜 모델링을 구현하여야 하며, CRO 등 여러 기관에서 생성된 기초 시험자료를 통합하여 저장하는 시스템을 국가적으로 갖추어야 할 것으로 생각된다.
Conclusion
인공지능 기술은 독성병리 분야에서 이미지 기반 진단, 질병 예후 예측과 위해성 평가 등에 사용되고 있다. 최근 인공지능의 발달이 가속화함에 따라 병변을 해석하는데 인공지능의 성능이 훈련된 독성병리전문가의 성능과 비교할 수 있는 정도인 것으로 나타났다. 인간과 인공지능 파트너십은 이미 많은 의료 환경에서 나타나고 있으며, 컴퓨터과학과 병리학의 협력은 앞으로 더 많이 이루어질 것으로 보인다. 하지만 현재의 시점으로 보아 인공지능 단독으로 병변을 판독하고 진단하는데 있어서 의료적 측면과 법률적 측면 등의 한계가 있으며, 병리전문가의 확인을 통해 정확도 등을 더 높여서 이러한 한계를 극복하여야 할 것으로 생각된다. 진단 결과에 대한 신뢰도 부족, 실용화 불편과 기능 단순성 등의 여러 한계를 인공지능 연구자, 소프트웨어 엔지니어와 의료전문가 사이에 긴밀한 협업으로 극복할 수 있다고 생각되고 있다[56].
인공지능 전용 독성병리학의 시작은 아직은 먼 것으로 보이며, 현 시점으로 볼 때 훈련된 독성병리전문가와 인공지능이 결합하여 진단을 진행할 경우, 인간 또는 인공지능 단독에 비해 진단 오류를 줄여줄 것으로 기대된다. 따라서 인공지능이 독성병리전문가의 개인 및 집단 진단 효능과 역량을 대체하는 것으로 생각할 것이 아니라, 독성병리전문가가 인공지능을 적극적으로 활용하여 진단의 정확성을 높이고, 병변 평가기술의 발전을 도모하고, 독성병리 관련 빅데이터 구축을 통해 신약 등의 효능과 안전성 평가에 정밀성을 높여야 한다고 생각한다.