Original Article
Database construction of small–molecule inhibitors as bovine viral diarrhea virus (BVDV) RNA-dependent RNA polymerase: a cheminformatics approach
Han-Ha Chai1,*, Bong-Hwan Choi2, Jong-Eun Park2, Ui-Hyung Kim3, Yong-Min Cho4, Dajeong Lim2,*
1National Institute of Animal Science, RDA, Wanju, 55365 and College of Pharmacy, Chonnam National University, Kwangju, 61186, Korea
2National Institute of Animal Science, RDA, Wanju, 55365, Korea
3Hanwoo Research Institute, National Institute of Animal Science, RDA, Pyeongchang, 25340, Korea
4Rural Development Administration, Wanju, 55365, Korea
*Corresponding author: Dajeong Lim, National Institute of Animal Science, RDA, Wanju, 55365, Republic of Korea +82-63-238-7306, +82-63-238-7347,
lim.dj@korea.krHan-Ha chai, National Institute of Animal Science, RDA and College of Pharmacy, Chonnam National University, Kwangju, 61186, Korea +82-63-238-7307, +82-63-238-7347,
hanha@korea.kr
© Research Institute of Veterinary Medicine, Chungbuk National University. All rights reserved.
Received: Feb 14, 2017; Accepted: Mar 15, 2017
Abstract
Bovine viral diarrhea virus (BVDV) is a major pathogen that may be one of the main reasons for economic losses in the livestock industry. BVDV is a well-characterized member of Flaviviridae family with plus-stranded RNA viruses. Non-structural NS5B protein is RNA-dependent RNA polymerase, which is responsible for viral RNA synthesis and genome replication of BVDV. Therefore, the NS5B polymerase is a key target for the discovery of anti-BVDV drugs. A number of small-molecule inhibitors against the NS5B polymerase have been reported in literature of which we collected series molecules having various scaffold with their biological data determined by evident experimental conditions, methods and procedures. Then, we constructed database of 655 small-molecule NS5B inhibitors having definitive activity values, structural parameters, and physicochemical properties (such as molecular hydrophobicity, hydrophilicity, polarity, Hbond donors and H-bond acceptors) associated with their absorption and permeability through a cheminformatics approach. The database was opened to provide insight for allosteric NS5B inhibitors of BVDV with an accessible platform on the web (http://nabic.rda.go.kr/chemical genomic database/BVDV RNA dependent RNA polymerase inhibitors). This molecular information in the database would be useful in attempting to identify features and decision factors that enhance anti-BVDV activity or increase selectivity of the allosteric inhibitor. These anti-BVDV molecules could also be screening for the purpose of exploiting potent NS5B inhibitors in the same family (e.g., HCV, CSFV, YFV, WNV, and DENV).
Keywords: BVDV; NS5B polymerase; allosteric inhibitor; cheminformatics; database
Introduction
가축 바이러스성설사병은 2017년 국제수역사무국(OIE)에 서 정하는 사회 및 국가 내 공중보건의 중요성이 인지된 116 개 가축질병리스트에 포함되어 있다[1]. 소에서 바이러스 성설사병을 일으키는 주요병원체는 소바이러스성설사병 바 이러스(Bovine Viral Diarrhea Virus, BVDV)이다. 특히 BVDV에 감염된 소에서는 감염시기와 바이러스 유전자형에 따라 유산등과 같은 번식문제, 우유생산량과 유품질 저하, 성장지체, 2차 세균감염, 송아지 폐사 등 매우 넓은 임상증 상이 나타난다. 또한, BVDV를 지속적으로 배출하는 지속감 염 송아지 발생과 면역저하에 따른 점막형 소바이러스설사 병 악화로 인하여 축산농가의 경제적 손실은 더 증폭된다. 현 재, BVDV의 근본적 치료제는 개발이 되어 있지 않으며, 축 산농가의 사양관리개선, 면역학적 예방관리(초유급여에 의 한 수동면역이나 사독혼합백신을 접종하여 능동면역 확보) 와 소바이러스설사병 조기발견 등 지속적인 예방 및 질병관 리가 최우선이다.
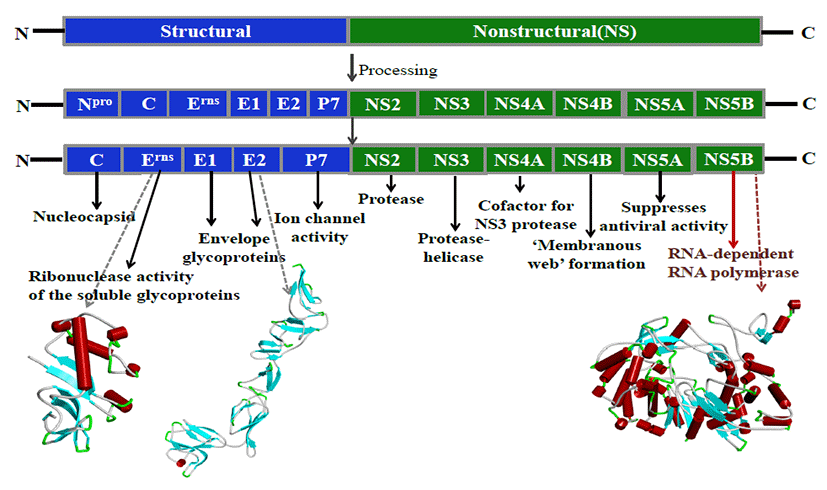
BVDV가 숙주세포에 부착 및 침투하여 바이러스 복제하는 과정은 10시간에서 20시간이 걸리지만, 백신에 의한 항체 가 생성되기 위해서는 최소한 10일에서 14일이 소요된다 [2-4]. 백신에 있는 BVDV 유전자형과 다른 변이유전자 형에 감염되면 예방효과가 낮아질 수 있기 때문에, 백신 단 독으로 예방하기보다는 직간접적으로 능동면역반응의 효과 를 더 증가시킬 수 있는 면역증강제가 병용요법(combination therapy)으로 적용되고 있다[5-7]. BVDV는 동일한 플라비바이러스과(Flaviviriridae)에 속하는 인간C형간염 바이러스(HCV)의 모델바이러스[8]로 숙주세포에서 감염 기전 및 공통된 분자적 특성을 가지는 단일가락의 RNA바이 러스(12.6kb의 유전자크기를 가지는 plus-stranded RNA genome)이다[3]. BVDV의 5개 구조단백질(뉴클레오캡시 드; C, BVDV-피막의 당단백질; Erns, E1, E2와 P7; 이온 채널관련 단백질)은 N-말단영역에 존재하는 반면에, 6개 비구조단백질(NS2, NS3, NS4A, NS4B, NS5A, NS5B) 은 C-말단영역에 위치한다[9]. 즉, BVDV 단백질서열은 NH2-Npro-C-Erns-E1-E2-P7-NS2-NS3-NS4ANS4B- NS5A-NS5B-COOH를 가진다(Fig. 1).
Fig. 1.
The BVDV genome is translated into a single polyprotein, which is processed into at least five structural and six nonstructural proteins for BVDV viral assembly and replication.
Download Original Figure
BVDV 제시(processing)과정은 N-말단영역의 Npro (autoprotease)에 의해 구조/비구조단백질 분해과정부터 시작된다. 숙주감염세포에서 BVDV 증식은 바이러스 부착 (attachment), 침입(penetration), 합성(biosynthesis), 조립(assembly and packing)의 과정을 거쳐 복제된 바 이러스를 방출(release)한다. 위와 같은 일련의 바이러스 증식과정을 상세하게 살펴본다면, BVDV 피막의 당단백질 (Erns, E1, E2)이 숙주세포표면의 항원제시 수용체(CD46, CD81)에 결합하여 숙주세포로 침입한다[10-13]. 숙주 내 단백질분해효소에 의해 뉴클레오캡시드(단백질 성분) 가 분해되어 바이러스 RNA는 감염세포로 방출된다. 숙주 세포 내로 방출된 BVDV의 단일가락 RNA(plus-stranded RNA)는 NS5B 리보핵산합성효소(RNA-dependent RNA polymerase, RdRp)가 상보적인 RNA가락(minusstrand) 을 합성할 때 주형(template)로 작용한다. 주형에 상보적인 RNA 가락(minus-strand)은 바이러스의 mRNA 로 작용하여 새로운 plus-stranded RNA를 합성하게 한다. 이 과정에서 minus-strand RNA는 double-strands RNA 중간체(intermediate)에만 존재하는 상보적인 RNA 가락 이다[30]. 동시에, 뉴클레오캡시드(숙주의 세포질에서)와 바이러스 피막에 삽입되는 당단백질(숙주의 소포체에서)이 합성된다. 합성된 BVDV 피막의 당단백질이 숙주의 소포를 통하여 세포막으로 운반되면, 뉴클레오캡시드가 조립되면서 새로 생성된 바이러스 RNA를 둘러싼다. 이렇게 뉴클레오캡 시드에 둘러싸인 바이러스 RNA는 숙주감염세포에서 분리 되어 나오면서, 바이러스 피막의 당단백질은 숙주의 소포체 에서 유래한 막에 삽입된다[13].
숙주 내, BVDV에 대한 면역방어반응은 바이러스 RNA를 인지하는 톨-유사수용체(TLR3, TLR7/TLR8) 매개 제1 형 인터페론(IFN-β)과 염증성사이토카인 NF-κB을 활 성화한다[14-17]. 특히, 비구조단백질의 변이는 숙주감 염세포의 세포변성효과(cytopathic effect, CPE)를 나타 나게 하는 중요한 원인으로 알려져 있다[18-20]. NS4B 는 integral membrane protein으로 숙주감염세포 내 골지 체와 미토콘드리아 막에 결합하여 숙주유래 단백질의 재배 열을 유도하거나 BVDV의 NS5A와 NS5B를 membranebound RNA replication complex로 조립하는 데 관여한다 [21]. NS5A는 숙주감염세포의 소포체에서 단백질 이동과 NF-κB 신호전달에 관여하는 NIBP(NIK- and IKKβ- binding protein)와 상호작용을 하는 데, 과발현된 NS5A 는 NIBP에 의한 NF-κB 활성화를 억제시킨다[22-23]. 숙 주의 감염세포의 변성을 유도하는 NS4B와 NS5A와 달리, NS5B 리보핵산합성효소는 바이러스 RNA를 합성할 때 자 체적으로 오류를 교정하는 기능이 없어 많은 변이를 유발한 다[24-25]. 더 나아가서, BVDV는 바이러스 유래 단백질 분해효소와 숙주 내 단백질분해효소를 이용하여 비구조단백 질를 분해할 수 있는데, 이때 다양한 바이러스 변이(viral or cellular insertion, deletion, single point mutation 등)가 생성된다. 예를 들어, 바이러스 유래 단백질분해효소 NS2 (cysteine-autoprotease)는 바이러스 감염초기에 NS2/ NS3 연결부위의 결합을 분해하여 NS3의 발현에 직접적 영 향을 준다[62]. NS3 (helicase and protease)의 serine protease domain은 NS3/NS4A, NS4B/NS5A, NS5A/ NS5B 연결부위의 Leu-Ser결합을 자르고 NS4A/NS4B 에서는 Leu-Ala결합을 분해한다. 이와 같이, 바이러스 유 래 단백질분해효소 NS2 와 NS3는 특정한 비구조단백질의 연결부위를 인식하여 cysteine 또는 serine 결합을 분해하 지만, 숙주 유래 단백질분해효소를 이용하면 비특이적으로 비구조단백질의 연결부위를 분해하도록 유도한다. 숙주세포 변성효과와 바이러스 자체의 변이를 통하여, BVDV의 비구 조단백질은 염증성사이토카인 NF-κB 활성화를 억제시키 지만, 구조단백질 Npro와 Erns, E2는 주로 제1형 인터페론 분비를 억제시킴으로써 숙주세포의 면역반응을 방해하는 것 으로 알려져 있다[61].
실험적으로 단백질 3차-구조가 결정된 BVDV의 피막 당단백질 중 Erns (PDB ID code: 4DVK, 4DVL, 4DVN, 4DW3, 4DW4, 4DW5, 4DW7, 4DWA)[27] 와 E2 (PDB ID code: 4JNT, 4ILD, 4DVK) [28], 그리고 NS5B 리보 핵산합성효소(PDB ID code: 1S48, 1S49, 1S4F, 2CJQ) [9,29-30]는 anti-BVDV효과를 가지는 후보물질을 탐 색하는 데 목표단백질이다(Fig. 1). 특히, NS5B 리보핵산 합성효소는 바이러스 RNA 합성과정에 직접적으로 관여하 고 있으며 동일한 플라비바이러스과에 속하는 HCV[31- 33], 뎅기열 바이러스(DENV)[34-35], 일본뇌염바이러 스(JEN)[36]에서도 NS5 리보핵산합성효소의 단백질 3 차-구조가 알려져 있기 때문에, RNA 바이러스의 저감물 질(inhibitor) 개발에 주요연구대상이다. 알려진 NS5 리보 핵산합성효소의 단백질 3차-구조 기반 저감물질(structure- based drug design)을 탐색하여, 플라비바이러스과 에 속하는 RNA 바이러스에 공통적으로 작용할 수 있는 비 특이적 [37-41] 또는 BVDV에만 특이적으로 작용할 수 있는 allosteric inhibitors를 발굴하는 연구가 많이 진행되 고 있다[42-55]. 그 중에서 BVDV의 NS5B 리보핵산합 성효소에 대한 저감물질은 염기성을 가지는 방향족 유도체 (arylazoenamine[42-43], imidazopyridine[44-45], benzimidazole[46], acridine[47]등)에서 연구되고 있다. 또한, 발굴된 저감물질은 비특이적 항바이러스제를 개발하 는 데, NS5B 리보핵산합성효소의 작용점(active site) 유 사성 정도에 따라 선도물질(lead compound)이 될 수 있 다. 한 예로, Arylazoenamine계열의 저해물질은 BVDV 이 외에도, 콕삭키바이러스-B2(CVB-2), 호흡기세포융합바 이러스(RSV)에도 항바이러스 효과를 가진다[42]. 다른 측 면에서, BVDV 저감물질들은 숙주 내 BVDV의 면역방어를 고려하여 톨-유사 수용체(TLR3, TLR7/TLR8)의 길항제 (agonists)로 작용할 수 있는 지를 탐색한다[48].
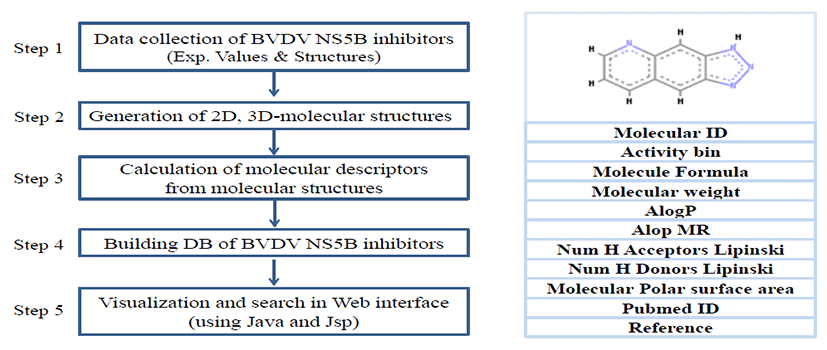
본 연구에서는 문헌 등에서 보고된 BVDV의 NS5B 리보 핵산합성효소에 대한 저감물질(분자량1300이하) 정보를 수집하였고, 이중에서 동일한 MTT 실험방법(measuring cell viability/cytotoxicity of compounds by tetrazolium dye)으로 BVDV의 NS5B 리보핵산합성효소에 대한 저해활 성(EC50, effective concentration in μM to achieve 50% protection of MDBK cells from BVDV-induced cytopathogenicity) 이 측정된 후보물질만을 선별하였다. 이렇게 선별된 후보물질들은 정확한 저감활성(EC50)값을 가지고 있 으며 다양한 유도체(basic aromatic scaffolds 이외 indole derivatives[49], γ-carboline derivatives[50], thiosemicabarzone[51], diphenylmethane[52-53], aromatic cationic molecules[54-55])를 가진 655개의 화 합물이다. 655개의 후보물질을 대상으로 3차원으로 분자구 조화와 3차원-분자구조로부터 신체 내 흡수(absorption) 와 세포막 투과(permeability)에 관련된 물리화학적 성질 들(분자의 크기, 분자의 소수성, 분자의 친수성, 수소결합 주 개수, 수소결합 받개수)을 계산하였다. 그리고, 실험적으로 측정된 저감활성값을 pEC50 (-logEC50)로 전환하여, 이를 Activity bin값으로 분류하였으며, Activity bin값이 클수록 저감활성이 큰 후보물질이다. 부가적으로, 후보물질이 발표 된 참고문헌정보(pubmed ID 등)도 포함하여 데이터베이스 를 구축하였다(http://nabic.rda.go.kr/chemical genomic database/ BVDV RNA dependent RNA polymerase inhibitors). 위와 같은 정보를 담은 데이터베이스를 사용자가 웹에서 특정한 범위 내에서 해당 데이터를 검색할 수 있도 록 하였으며, 검색된 결과 내에서 추가 검색(‘and’검색) 이 가능하도록 구현하였다. 이를 통해 655개 후보물질들이 공통적으로 가지고 있는 substructure 기반 물리화학적 성 질들의 상세정보를 제공한다.
Materials and Methods
BVDV의 NS5B 리보핵산합성효소에 대한 저감물질의 특성 을 해석하기 위하여 선별된 655개의 후보물질에 대한 분자 구조를 Accerlys사의 Drug discovery studio (DS) 프로그 램의 visualizer 모듈을 사용하여 3차원으로 분자구조화 및 이를 SD파일(각원자의 위치좌표 및 결합형태 등 정보포함) 로 저장하였다. 이는 후보물질의 3차원-구조로부터 분자표 현자(molecular descriptor, 화학구조의 특징을 수치로 표 현함)를 계산하기 위함이다. 동시에 사용자가 웹에서 분자 구조 정보를 쉽게 볼 수 있도록, 각각의 후보물질마다 2차 원-분자구조로 변환하여 이미지파일(PNG파일형태)로 저 장하였다. 같은 분자량을 가진 후보물질이라도 다른 분자식 (molecular formula)을 가질 수 있기 때문에, 각 후보물질 마다 ID를 부여하면서(NMID0001부터 NMID0655까지), 해당 후보물질의 분자식 정보도 추가하였다. 다음단계로, 각 후보물질마다 분자의 구조적 특성으로부터 신체 내 흡수와 세포막 투과에 관련된 물리화학적 특성들을 DS의 calculate molecular properties 모듈에서 6개의 분자표현자(분자량, AlogP, AlogP-MR, Molecular polar surface area, Num H-acceptors Lipinski, Num H-donors Lipinski)로 수 치화하였다. 계산된 분자표현자중에서 AlogP와 AlogPMR[56]은 분자의 소수성(hydrophobicity)를 측정하는 척 도이다. AlogP는 두 용매(옥탄올과 물)로 구성된 한 개의 시 스템에서 분자의 분배계수(logP(octanol-water))를 적용한다.(식1)
여기서 ni는 I형태의 원자개수이고, ai는 I형태의 원자가 기 여하는 소수성 값이다. AlogP (atomic level hydrophobicity as log of the octanol-water partition coefficient) 는 분자의 소수성 측정을 서로 혼합되지 않는 한 개의 용 매시스템에서 상대적 분배계수로 친수성/소수성에 해당하 는 지를 나타내는 반면에, 아래의 식2에 따라 AlogP-MR (hydrophobicity with molar refractivity)은 AlogP에 분 자의 부피(molecular volume)와 편극성(molecular polarizability) 에 대한 정보를 부여한 것이다.
한 개의 분자 내에서 ni는 I형태의 원자개수이고, bi는 I형 태의 원자가 기여하는 molar refractivity값인데, molar refractivity=4пNα/3 이고 여기서 N은 Avogadro수 이 며, α는 편극성이다. 만일 분자가 구형의 형태를 가진다 면, 편극성은 분자반지름의 세제곱(α=r3)과 같아서, molar refractivity=4пr3/3로 분자의 부피와 같다. 예를 들 어, 한 개의 분자 내에서 편극성을 띤 영역이 넓으면 molar refractivity도 커지고 AlogP-MR값도 증가한다. AlogPMR와 같이, 분자의 편극성과 연관성이 있는 molecular polar surface area(PSA) 분자표현자는 3차원-분자구조 에서 극성을 가지는 작용기(functional group)가 물에 노출 된 표면적의 합이다[57].(식3)
여기서 ntypes는 극성을 가지는 작용기 종류의 수이고, c(fragmenti)는 분자 내 I형태의 작용기가 기여하는 물에 노 출된 표면적에 상수 c의 곱이며, ni는 분자 내 I형태의 작용 기 빈도수이다. 이는 분자의 극성표면적이 크면 클수록, 신 체 내 물과 상호작용을 더 잘하여 친수성(hydrophilicity)도 커진다. 다른 측면에서 신체 내 흡수에는 물과 수소결합 할 수 있는 분자의 특징도 중요하기 때문에, 후보물질에서 수소 결합 주개(Num H-donors Lipinski, 수소를 줄 수 있는 질 소와 산소에 결합되어 있는 수소원자 개수의 합)와 수소결 합 받개(Num H-acceptors Lipinski, 수소를 받을 수 있는 비공유 전자쌍을 가지는 질소와 산소원자 개수의 합)로 수 소결합과 관련된 2개의 분자표현자를 계산하였다. BVDV NS5B 리보핵산합성효소의 저해물질을 데이터베이스로 구 축하는 단계와 제공되는 655개 후보물질의 상세정보를 Fig. 2에서 도식화하였다.
Fig. 2.
Schematic diagram of database for 655 inhibitors against NS5B RNA polymerase of BVDV and its detail information of an inhibitor provided in the database.
Download Original Figure
위와 같이 구축된 BVDV의 NS5B 리보핵산합성효소에 대 한 저해물질의 상세정보를 사용자가 웹 인터페이스에서 볼 수 있도록 공개하였다. 웹인터페이스를 구현하는데 Java 프 로그래밍 언어와 JSP (JavaServer Pages)웹프레임워크가 사용되었다. 사용자는 메인메뉴를 통해 테이블 형태의 요약 정보를 확인하거나, 돋보기모양의 아이콘을 클릭하여 후보 물질마다 분자구조가 포함된 10개의 상세정보를 확인할 수 있다. 또한, 특정범위 내에 존재하는 후보물질의 물리화학적 특성 등을 조건검색 할 수 있도록 구현하였다[Fig. 4참조].
Results
NS5B 리보핵산합성효소의 저감물질 특성
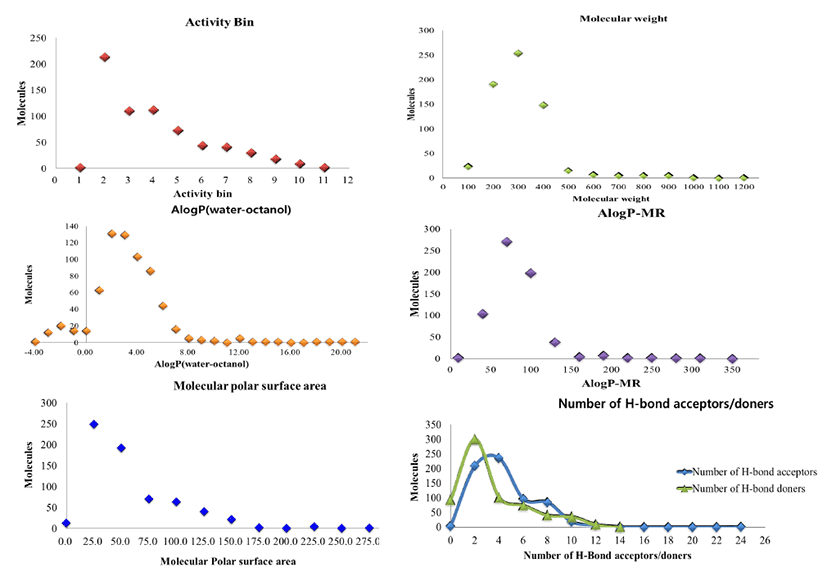
선별된 BVDV의 NS5B 리보핵산합성효소에 대한 655개 저 감물질들 중 85%이상은 분자량이 500이하(632개)인 분자 들이고, 소수성의 물리화학적 특성이 AlogP≤5.0, AlogPMR ≤100 범위에 해당하는 분자는 573개이다. 또한, 574 개 후보물질들은 분자 내 극성표면적이 25.0 Å2≤3D PSA ≤ 100.0 Å2범위에 존재하며, 수소결합 주개(≤5개)와 수 소결합 받개(≤10개)를 만족하는 후보물질도 640여개가 넘 는다. 아래의 Fig. 3은 655개 후보물질이 갖고 있는 물리화 학적 특성들에 대한 6개 분자표현자 값의 분포를 나타낸 것 이다.
위에서 언급한 6개 분자표현자는 작은 분자량(1300이하) 을 가지는 유효물질이 세포막(친수성/소수성 특성을 가짐) 을 통과하여 신체 내에 흡수되는 정도를 설명하는 물리화학 적 특성과 관련성이 있다. 이런 특성들은 화이자제약의 부 사장이였던 Lipinski 박사가 임상시험후보물질들의 druglike properties을 제시한 ‘Lipinski's rule of five’를 반 영한 것이다[58-60]. 예를 들어, 후보물질의 AlogP값이 너 무 크면 지질에는 잘 녹을 수 있지만, 경구 흡수가 어렵다. 반대로, 분자의 극성표면적(3D PSA)또는 AlogP-MR값이 너무 크면, 위와 장의 세포벽 통과가 안되고 더욱이 간의 대사과정시스템에 의해 분해되어 유효성이 감소하게 된다.
‘Lipinski's rule of five’에 따라[58-59], 구축된 NS5B 리보핵산합성효소의 저해물질 42.4%이상(277개)은 경구 흡수(orally absorption)와 세포막통과(permeability)와 밀접한 관계가 있는 6개 분자표현자의 범위(분자량이 500 이하, AlogP≤5.0, AlogP-MR ≤100, 25.0 Å2≤3D PSA ≤ 100.0 Å2, H-donors ≤5, H-acceptors ≤10)를 동시 에 만족시킨다. 이로부터 분자표현자의 범위를 만족시키는 42.4%이상의 후보물질들이 모두 선도물질이 될 수 있는 것 은 아니지만, NS5B 리보핵산합성효소의 작용점 특성과 저 감물질의 구조- 활성간의 관계(structure-activity relationship) 정보와 함께 후보물질의 우선순위를 정렬(scoring) 하는데 filtering 기준이 될 수 있다.
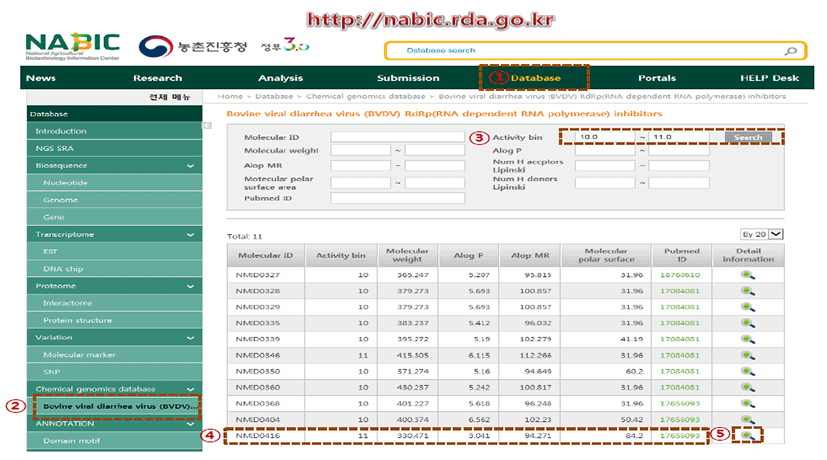
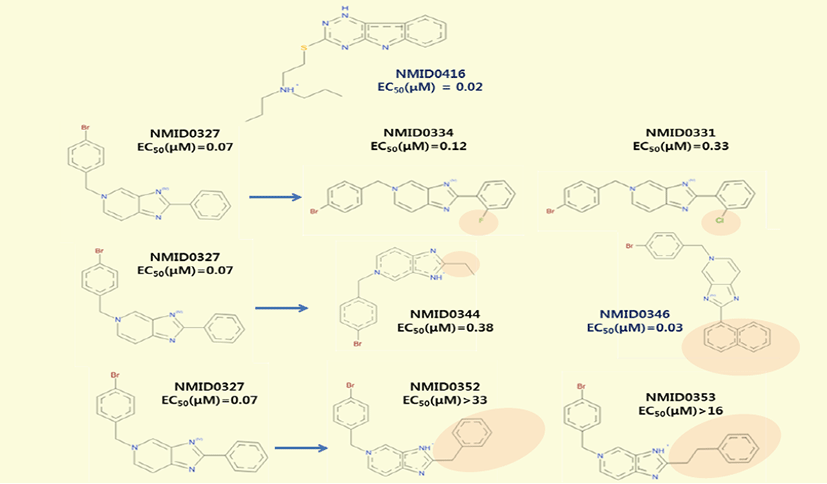
한편, 물리화학적 특성을 고려하지 않고 저감활성이 높은 후보물질들을 검색하고자 한다면, Fig. 4와 같이 Activity bin값을 10에서 11까지 범위를 정하여 조건검색으로 11개 의 후보물질을 확인할 수 있다. 그 중에서 저감활성이 높 은 후보물질(Activity bin값이 11임)은 NMID0346[66]과 NMID0416[63]이다.
Fig. 4.
Searching data in BVDV RdRp inhibitors and viewing result by inputting a query entry (10 ≤Activity bin≤11) in search box. The result was displayed in a separate table.
Download Original Figure
NS5B 리보핵산합성효소의 저감물질 정보분석 예시: 저감활성이 큰 NMID0346
경구흡수와 세포막통과와 연관된 6개의 분자표현자 의 범위를 모두 만족시키면서 BVDV의 NS5B 리보핵 산합성효소에 대한 저감활성이 가장 높은 후보물질은 NMID0416 (EC50 (μM)=0.02, Activity bin값이 11임) 이다. NMID0416은 VP32947로 잘 알려진 BVDV의 저 해물질로서 3-[((2-dipropylamino)ethyl)thio]-5H- 1,2,4-triazino[5,6-b]indole이고, 또 다른 저감활성이 큰 NMID0346은 2,5-disubstituted imidazo[4,5-c] pyridine계열의 후보물질이다. NMID0416은 BVDV에 대 한 특이적으로 작용하는 저해물질인 반면에 NMID0346와 같은 imidazo[4,5-c]pyridine계열의 후보물질들은 BVDV 이외에 CSFV(classical swine fever virus)의 NS5B 리 보핵산합성효소에도 저감활성을 가지고 있다[68]. 구축 된 anti-BVDV 후보물질의 데이터베이스의 활용예제로서 NMID0346처럼 Imidazo[4,5-c]pyridine 유도체의 구조 를 가지고 있으면서 2,5-위치에서 다른 작용기가 존재하 는 89개 후보물질들(NMID0327~NMID0415)을 이용하 여 저감활성에 영향을 미치는 치환기 효과를 분석하고자 한 다[Fig. 5참조]. Imidazo[4,5-c]pyridine에서 2번위치에 phenyl ring이 도입이 되고, 동시에 5번 위치에 benzyl로 치환이 되면(5-benzyl-2-phenyl-5H-imidazo[4,5- c]pyridine, EC50 (μM)=0.25) BVDV의 NS5B 리보핵 산합성효소 저감활성이 더 커진다[69]. 더 나아가서 5번 위치에 4-bromobenzyl 치환(NMID0327)하면 저감활 성(EC50 (μM)=0.07, Activity bin값이 10임)이 증가한 다. NMID0327(5-[4-bromobenzyl]-2-phenyl-5Himidazo[ 4,5-c]pyridine)에서 2번위치에 2-halophenyl 기로 치환하면(NMID0331~NMID338) 저감활성은 약간 감소된다(EC50 (μM)=0.12~0.5, Activity bin값의 범위는 7~10를 가짐). 2번위치의 phenyl기 대신에 2-naphthyl기 (NMID0346, EC50 (μM)=0.03, Activity bin값이 11임)가 도입되면 저감활성이 NMID0327보다 약간 증가한다. 그러 나 2번위치의 phenyl기 대신에 benzyl기(NMID0352, EC50 (μM)=33 Activity bin값이 3임) 또는 2-phenylethyl기 (NMID0353, EC50 (μM)=16, Activity bin값이 4임)로 치 환되면 저감활성이 많이 줄어든다. 이는 imidazo[4,5-c] pyridine계열의 후보물질들이 결합하는 BVDV의 NS5B 리 보핵산합성효소의 작용점의 크기와 상호작용하는 아미노산 의 물리화학적 특성이 간접적으로 반영이 된 것이다.
Fig. 5.
Structure activity relationship of the anti-BVDV NS5B polymerase of 5-[4-bromobenzyl]-2-subsituted-5Himidazo[ 4,5-c]pyridines compared with NMID0327. The EC50 is the 50% effective concentration against BVDV-induced cytopathic effect [66-68].
Download Original Figure
NS5B 리보핵산합성효소의 작용점
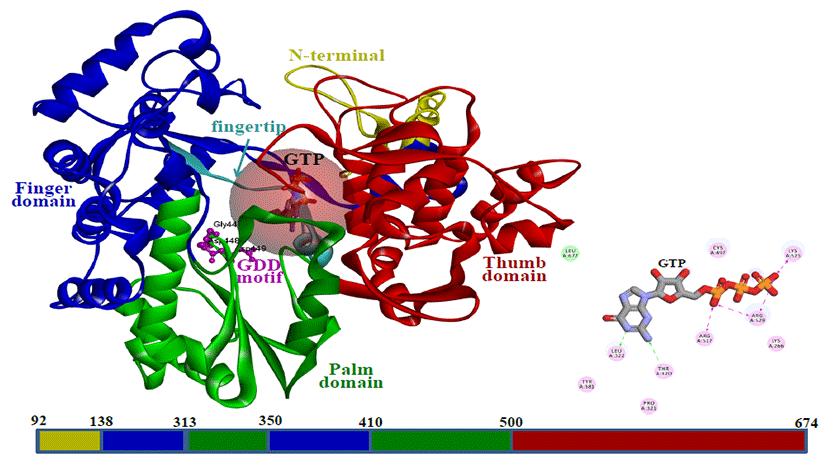
BVDV의 NS5B 리보핵산합성효소는 720개 아미노산으로 구성되어 있지만, 3차원-단백질구조는 Val92부터 Leu679 까지 영역에서만 X-ray 실험방법으로 결정되었다. NS5B 리보핵산합성효소의 3차원-단백질구조는 N-말단영역의 일부분(Val92-Gln138)을 포함하여 right-hand 형태로 배열된 3개 도메인(Finger, Palm, and Thumb domains) 이 작용점을 둘러쌓고 있다(Fig. 6참조). 특히, fingertip (Ala260-Gln288)와 N-terminal 영역(Val92-Gln138), Thumb 도메인의 C-말단영역 loop(GLN670-Leu679)은 작용점의 출구역할을 하며, NS5B 리보핵산합성효소가 새로 운 상보적인 바이러스 RNA가락(임시적으로 존재하는 minus- stranded RNA 중간체로부터 새로운 plus-stranded RNA)을 합성할 때 이들의 conformer가 유동적으로 바뀌 어서 더 커진 작용점의 공간(cavity)을 제공하게 된다. 특히, 이 영역은 BVDV와 HCV의 NS5B 리보핵산합성효소의 3 차-단백질 구조에서 작용점의 크기와 상호작용하는 아미노 산 특성의 차이를 나타내게 한다. 다만, fingertip은 Finger 와 Thumb 도메인을 연결하는 영역으로 conformer가 바뀌 어도 작용점의 공간은 제한적일 수 밖에 없다. NS5B 리보 핵산합성효소는 주형이 되는 plus-stand RNA이외에 기질 (substrate)인 GTP(Guanosine-5'-triphosphate)와 새 로 합성된 바이러스 RNA의 triphosphate를 안정화 시킬 수 있는 양이온(Mg2+ 또는 Mn2+)과 Palm도메인에 있는 Gly447-Asp448-Asp449(GDD) motif와의 상호작용을 필요로 한다. NS5B 리보핵산합성효소의 기질인 GTP-결합 부위(GDD motif의 6Å내)는 Lys266, Thr320, Pro321, Leu322, Cys446, Cys497, Ser498, His499, Arg517, Lys525, Arg529, Tyr581, Leu677로 구성되어 있다. NS5B 리보핵산합성효소 작용점 내에 있는 GTP-결합부위 와 상호작용하는 아미노산의 정보를 Fig. 6에서 나타냈다.
Fig. 6.
The structure of BVDV NS5B polymerase-GTP complex and the GTP-binding site. The N-terminal (residues 92-138), finger (residues 139-313 and 351-410), palm (residues 314-350 and 411-500), and thumb (residues 501-674) domains are colored by yellow, blue, green, and red, respectively. The fingertip region (residues 260-288) is shown in cyan.
Download Original Figure
BVDV의 NS5B 리보핵산합성효소에서 주형인 plus-stand RNA 가락은 작용점에서 GDD motif에서 약 4Å내 인접해서 위치[10,29-30]하여 Palm 도메인과 상호작용(Ala341- Asp345, Asp448-Leu452, Ala463, Lys486-Ala488, and Glu495-His499)을 하는 반면에, 상보적인 minusstand RNA 가락은 GTP의 위쪽에 있어 N-말단영역 (Asn136)과 Thumb 도메인(Arg 517, Val521-Ile522, Lys525-Arg529, Phe549, 그리고 Thr580)과 상호작용 을 하게 된다. 현재까지 BVDV의 NS5B 리보핵산합성효소 와 주형인 바이러스 RNA 또는 저감물질간의 결합구조는 알 려져 있지 않다. 655개의 후보물질들 중에서는 allosteric inhibitor로 작용한다면, 작용점 이외에 다른 영역에 결합 하여 NS5B 리보핵산합성효소를 저감할 수 있다. 알려져 있 는 allosteric inhibitor인 NMID0436경우에는 NS5B 리보 핵산합성효소의 작용점에서 약 20Å 떨어진 Finger 도메인 의 Phe224를 중심으로 Ile390, Ala392, 그리고 Leu225 와 상호작용을 하면서 Finger 도메인과 fingertip 영역의 유동성을 제한하면서 저감하는 것으로 알려져 있다[63]. NMID0436와 유사하게 NMID0346도 Phe224를 중심으 로 Ala221, Ala222와도 상호작용[64]을 하는 반면에, 또 다른 CF02334 allosteric inhibitor인 3-(imidazo[1,2- a:5,4-b']dipyridin-2-yl)aniline은 Asn264를 중심으로 fingertip 영역에 있는 Arg157과 상호작용을 하는 것으로 알려져 있다[65]. 본 연구에서 구축한 데이터베이스 내에 있 는 BVDV의 NS5B 리보핵산합성효소 저감물질의 상세정보 에 allosteric inhibitors들이 결합할 수 있는 부위와 GTP기 질이 인접해 있는 작용점의 특성을 고려하여 후보물질들에 서 요구되는 물리화학적 특성들에 얼마나 부합되는 지에 대 한 적합도 계산은 향후에 진행될 예정이다.
Conclusion
BVDV의 NS5B 리보핵산합성효소에 대한 저감물질의 정 보를 수집하여 동일한 MTT 실험방법으로 저감활성이 측정 된 655개 후보물질을 선별하였고, 선별된 후보물질의 저감 활성, 2차원-분자구조, 분자식, 신체 내 흡수와 세포막 투과 에 관련된 6개 물리화학적 성질들(분자의 크기, 분자의 소 수성, 분자의 친수성, 수소결합 주개수, 수소결합 받개수), 참고문헌 정보 등을 포함하여 데이터베이스로 구축하였다. 구축된 정보는 웹페이지(http://nabic.rda.go.kr/chemical genomic database/BVDV RNA dependent RNA polymerase inhibitors)에서 조건검색이 가능하도록 하였고, 조 건검색의 결과를 테이블 형태의 간단한 정보 또는 1개의 후 보물질에 대한 상세정보를 확인할 수 있도록 하였다. 본 연 구에서 구축된 655개의 후보물질의 정보로부터 일차적인 구조-활성간의 관계분석을 할 수 있지만, 추가적으로 알려 진 BVDV의 NS5B 리보핵산합성효소의 3차원-단백질구조 (PDB ID code: 1S48, 1S49, 1S4F, 2CJQ)정보를 이용 하여 작용점의 정보로부터 결합력에 영향을 주는 decision factors을 더 고려해야 한다. 동시에 저감활성이 높은 분 자들의 공통된 물리화학적 특성정보 분석도 더 진행되어야 할 것이다. 이번 연구를 통하여 구축된 데이터베이스로부터 655개 후보물질의 분자구조와 BVDV의 NS5B 리보핵산합 성효소에 대한 저감활성간의 관계정보는 BVDV에 대한 숙 주면역반응을 고려하여 톨-유사수용체 8을 활성화할 수 있 는 유용물질을 탐색하는 데 활용된 적이 있다. 더 나아가서, BVDV이외에 동일한 플라비바이러스과에 속하는 다른 바이 러스(HCV, CVB-2, RSV, CSFV등)에도 비특이적으로 작 용하는 저감물질을 디자인 하는 데 응용될 예정이다.
Acknowledgements
본 연구는 농촌진흥청 국립축산과학원 연구사업 (PJ00871103)으로부터 연구비를 지원받아 수행하였으며, 연구비 지원에 감사드립니다.
References
The world Organisation for Animal Health (OIE).
http://www.oie.int/en/animal-helath-in-the-world/oie-listed-diseases-2017
. 2017
Gong Y, Trowbridge R, Macnaughton TB, Westaway EG, Shannon AD, Gowans EJ. Characterization of RNA synthesis during a one-step growth curve and of the replication mechanism of bovine viral diarrhea virus. J Gen Virol. 1996; 77:2729-2736.

Li Y, McNally J. Characterization of RNA synthesis and translation of bovine viral diarrhea virus (BVDV). Virus Genes. 2001; 23:149-155.

Kalaycioglu A. Bovine viral diarrhea virus (BVDV) diversity and vaccination. A review. Vet Q. 2007; 29:60-67.

Finkielsztein LM, Moltrasio GY, Caputto ME, CastroEF. Cavallaro LV, Monglioni AG. What is known about the antiviral agents active against bovine viral diarrhea virus (BVDV)?. Curr Med Chem. 2010; 17:2933-2955.

Newcomer BW, Walz PH, Givens MD. Potential applications for antiviral therapy and prophylaxis in bovine medicine. Anim Health Res Rev. 2014; 15:102-107.

Newcomer BW, Givens MD. Approved and experimental countermeasures against pestiviral diseases Bovine viral diarrhea classical swine fever and border disease. Antiviral Res. 2013; 100:133-150.

Buckwold VE, Beer BE, Donis RO. Bovine viral diarrhea virus as a surrogate model of hepatitis C virus for the evaluation of antiviral agents. Antiviral Res. 2003; 60:1-15.

Meyers G, Thiel HJ. Molecular characterization of pestiviruses. Adv Virus Res. 1996; 47:53-118.

Choi KH, Groarke JM, Young DC, Kuhn RJ, Young DC, Kuhn RJ, Smith JL, Pevear DC, Rossmann MG. The structure of the RNA-dependent RNA polymerase from bovine viral diarrhea virus establishes the role of GTP in de novo initiation. Proc Natl Acad Sci USA. 2004; 101:4425-4430.

Krey T, Himmelreich A, Heimann M, Menge C, Thiel HJ, Maurer K, Rümeanpf T. Function of bovine CD46 as a cellular receptor for bovine viral diarrhea virus is determined by complement control protein 1. J Virol. 2006; 80:3912-3922.

Fredericksen F, Delgado F, Cabrera C, Yáñez A, Gonzalo C, Villalba M, Olavarría VH. The effects of reference genes in qRT-PCR assays for determining the immune response of bovine cells (MDBK) infected with the Bovine Viral Diarrhea Virus 1 (BVDV-1). Gene. 2015; 569:95-103.

Le Breton M, Meyniel-Schicklin L, Deloire A, Coutard B, Canard B, de Lamballerie X, Andre P, Rabourdin-Combe C, Lotteau V, Davoust N. Flavivirus NS3 and NS5 proteins interaction network a high-throughput yeast two-hybrid screen. BMC Microbiol. 2011; 11:234.

Sampath A, Padmanabhan R. Molecular target for flavivirus drug discovery. Antiviral Res. 2009; 81:6- 15.

Magkouras I, Mätzener P, Rümeanpf T, Peterhans E, Schweizer M. RNase-dependent inhibition of extracellular, but not intracellular, dsRNA-induced interferon synthesis by Erns of pestiviruses. J Gen Virol. 2008; 89:2501-2506.

Mätzener P, Magkouras I, Rümeanpf T, Peterhans E, Schweizer M. The viral RNase E(rns) prevents IFN type-I triggering by pestiviral single- and doublestranded RNAs. Virus Res. 2009; 140:15-23.

Peterhans E, Jungi TW, Schweizer M. BVDV and innate immunity. Biologicals. 2003; 31:107-111.

Luo D, Vasudevan SG, Lescar J. The flavivirus NS2B-NS3 protease-helicase as a target for antiviral drug development. Antiviral Res. 2015; 118:148-158.

Aleshin AE, Shiryaev SA, Strongin AY, Liddington RC. Structural evidence for regulation and specificity of flaviviral protease and evolution of the flaviviridae fold. Protein Sci. 2007; 16:795-806.

Frick DN, Lam AV. Understanding helicases as a means of virus control. Curr. Pharm. Des. 2006; 12:1315-1338.

Weiskircher E, Aligo J, Ning G, Konan KV. Bovine viral diarrhea virus NS4B protein is an integral membrane protein associated with Golgi markers and rearranged host membranes. Virol J. 2009; 6:185-199.

Tellinghuisen TL, Paulson MS, Rice CM. The NS5A protein of bovine viral diarrhea virus contains an essential zinc binding site similar that of the hepatitis C virus NS5A protein. J Virol. 2006; 80:7450-7458.

Zahoor MA, Yamane D, Mohamed YM, Suda Y, Kobayashi K, Kato K, Tohya Y, Akashi H. Bovine viral diarrhea virus non-structural protein 5A interacts with NIK and IKKbeta-binding proteins. J Gen Virol. 2010; 91:1939-1948.

Agopov EV, Murray CL, Frolov I, Qu L, Myers TM, Rice CM. Uncleaved NS2-3 is required for production of infectious bovine viral diarrhea virus. J Virol. 2004; 78:2414-2425.

Kameyama K, Sakoda Y, Matsuno K, Ito A, Tajima M, Nakamura S, Kida H. Cleavage of the NS2-3 protein in the cells of cattle persistently infected with non-cytopathogenic bovine viral diarrhea virus. Microbiol Immunol. 2008; 52:277-282.

Zahoor MA, Yamane D, Mohamed YM, Kobayashi K, Kato K, Tohya Y, Akashi H. Characterization and application of monoclonal antibodies to bovine viral diarrhea virus nonstructural protein 5A. Arch Virol. 2009; 154:1745-1754.

Krey T, Bontems F, Vonrhein C, Vaney MC, Bricongne G, Rümeanpf T, Rey FA. Crystal structure of the pestivirus envelope glycoprotein E(rns) and mechanistic analysis of its ribonuclease activity. Structure. 2012; 20:862-873.

Li Y, Wang J, Kanai R, Modis Y. Crystal structure of glycoprotein E2 from bovine viral diarrhea virus. Proc Natl Acad Sci USA. 2013; 110:6805-6810.

Choi KH, Gallei A, Becher P, Rossmann MG. The structure of bovine viral diarrhea virus RNA-dependent RNA polymerase and its amino-terminal domain. Structure. 2006; 14:1107-1113.

Choi KH, Rossmann MG. RNA-dependent RNA polymerase from Flaviviridae. Curr Opin Struct Biol. 2009; 19:746-751.

Mosley RT, Edwards TE, Murakami E, Lam AM, Grice RL, Sofia MJ, Furman PA, Otto MJ. Structure of hepatitis C virus polymerase in complex with primer-template RNA. J Virol. 2012; 86:6503-6511.

O'Farrell D, Trowbridige R, Rowlands D, Jäger J. Substrate complexes of hepatitis C virus RNA polymerase (HC-J4): structural evidence for nucleotide import and de-novo initiation. J Mol Biol. 2003; 326:1025-1035.

Biswal BK, Wang M, Cherney MM, Yannopoulos CG, Billimoria D, Bedard J, James MN. Non-nucleoside inhibitors binding to hepatitis C virus NS5B polymerase reveal a novel mechanism of inhibition. J Mol Biol. 2005; 361:33-45.

Rigat K, Wang Y, Hudyma TW, Ding M, Zheng X, Gentles RG, Beno BR, Gao M, Roberts SB. Ligandinduced changes in hepatitis C virus NS5B polymerase structure. Antiviral Res. 2010; 88:197-206.

Klema VJ, Ye M, Hindupur A, Teramoto T, Gottipati K, Padmanabhan R, Choi KH. Dengue virus nonstructural protein 5 (NS5):assembles into a dimer with a unique methyltransferase and polymerase interface. PLos Pathog. 2016; 19:e1005451.

Lim SP, Noble CG, Shi PY. The dengue virus NS5 protein as a target for drug discovery. Anntiviral Res. 2015; 119:57-67.

Lim SP, Wang QY, Noble CG, Chen YL, Dong H, Zou B, Yokokawa F, Nilar S, Smith P, Beer D, Lescar J, Shi PY. Ten years of dangue drug discovery progress and prospects. Anntiviral Res. 2013; 100:500-519.

Lu G, Gong P. Crystal structure of the full-length Japanese encephalitis virus NS5 reveals a conserved methyltransferase-polymerase interface. PLos Pathog. 2013; 9:e1003549.

Davidson AD. Chapter 2 New insights into flavivirus nonstructural protein 5. Adv Virus Res. 2009; 74:41-101.

Klema VJ, Padmanabhan R, Choi KH. Flaviviral replication complex coordination between RNA synthesis and 5'-RNA capping. Viruses. 2015; 7:4640-4656.

Wu J, Liu W, Gong P. A structural overview of RNAdependent RNA polymerase from the Flaviviridae family. Int J Mol Sci. 2015; 16:12943-12957.

Tonelli M, Boido V, Canu C, Sparatore A, Sparatore F, Paneni MS, Fermeglia M, Pricl S, La Colla P, Casula L, Ibba C, Collu D, Loddo R. Antimicrobial and cytotoxic arylazoenamines. PartIII: antiviral activity of selected classes of arylazoenamines. Bioorg Med Chem. 2008; 16:8447-8465.

Giliberti G, Ibba C, Marongiu E, Loddo R, Tonelli M, Boido V, Laurini E, Posocco P, Fermeglia M, Pricl S. Synergistic experimental/computational studies on arylazoenamine derivatives that target the bovine viral diarrhea virus RNA-dependent RNA polymerase. Bioorg Med Chem. 2010; 18:6055-6068.

Paeshuyse J, Letellier C, Froeyen M, Dutartre H, Vrancken R, Canard B, De Clercq E, Gueiffier A, Teulade JC, Herdewijn P, Puerstinger G, Koenen F, Kerkhofs P, Baraldi PG, Neyts J. A Pyrazolotriazolopyrimidinamine inhibitor of bovine viral diarrhea virus replication that targets the viral RNA-dependent RNA polymerase. Antiviral Res. 2009; 82:141-147.

Paeshuyse J Chezal JM, Froeyen M, Leyssen P, Dutartre H, Vrancken R, Canard B, Letellier C, Li T, Mittendorfer H, Koenen F, Kerkhofs P, De Clercq E, Herdewijn P, Puerstinger G, Gueiffier A, Chavignon O, Teulade JC, Neyts J. The imidazopyrrolopyridine analogue AG110 is a novel, highly selective inhibitor of pestiviruses that targets the viral RNA-dependent RNA polymerase at a hot spot for inhibition of viral replication. J Virol. 2007; 81:11046-11053.

Tonelli M, Boido V, La Colla P, Posocco P, Paneni MS, Fermeglia M, Pricl S. Pharmacophore modeling, resistant mutant isolation, docking, and MMPBSA analysis Combined experimental/computerassisted approaches to identify new inhibitors of the bovine viral diarrhea virus (BVDV). Bioorg Med Chem. 2010; 18:2304-2316.

Tonelli M, Vettoretti G, Tasso B, Novelli F, Boido V, Sparatore F, Busonera B, Ouhtit A, Farci P, Blois S, Giliberti G, La Colla P. Acridine derivativies as anti- BVDV agents. Antiviral Res. 2011; 91:133-141.

Chai HH, Lim D, Suk JE, Choi BH, Cho YM. Design of anti-BVDV drug based on common chemical features, their interaction, and scaffolds of TLR8 agonists. Int J Biol Macromol. 2016; 92:1095-1112.

Giampieri M, Balbi A, Mazzei M, La Colla P, Ibba C, Loddo R. Antiviral activity of indole derivatives. Antiviral Res. 2009; 83:179-185.

Sako K, Aoyama H, Sato S, Hashimoto Y, Baba M. Gamma- carboline derivatives with anti-bovine viral diarrhea virus (BVDV) activity. Bioorg Med Chem. 2008; 16:3780-3790.

Castro EF, Fabian LE, Caputto ME, Gagey D, Finkielsztein LM, Moltrasio GY, Moglioni AG, Campos RH, Cavallaro LV. Inhibition of bovine viral diarrhea virus RNA synthesis by thiosemicarbazone derived from 5,6-dimethoxy-1-indanone. J Virol. 2011; 85:5436-5445.

Salim MT, Okamoto M, Hosoda S, Aoyama H, Hashimoto Y, Baba M. Anti-bovine viral diarrhea virus activity of novel diphenylmethane derivatives. Antivir Chem Chemother. 2010; 20:193-200.

Hosoda S, Aoyama H, Goto Y, Salim MT, Okamoto M, Hashimoto M, Baba M, Hashimoto Y. Discovery of diphenylmethane analogs as anti-bovine diarrhea viral agents. Bioorg Med Chem Lett. 2009; 19:3157-3161.

Givens MD, Dykstra CC, Brock KV, Stringfellow DA, Kumer A, Stephens CE, Goker H, Boykin DW. Detection of inhibition of bovine viral diarrhea virus by aromatic cationic molecules. Antimicrob Agents Chemother. 2003; 47:2223-2230.

Givens MD, Galik PK, Riddell KP, Dykstra CC, Brock KV, Stringfellow DA. Effects of aromatic cationic molecules on bovine viral diarrhea virus and embryonic development. Theriogenology. 2005; 63:1984-1994.

Ghose AK, Crippen G. Atomic physicochemical parameters for three-dimensional structure-directed quantitative structure-activity relationships I Partition coefficient as a measure of hydrophobicity. J Comp Chem. 1986; 7:565-577.

Ertl P, Rohde B, Selzer P. Fast calculation of molecular polar surface area as a sum of fragmentbased contributions and its application to the prediction of drug transport properties. J Med Chem. 2000; 43:3714-3717.

Lipinski CA, Lombardo F, Dominy BW, Feeney PJ. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings. Adv Drug Deliv Rev. 2001; 46:3-26.

Veber DF, Johnson SR, Cheng HY, Smith BR, Ward KW, Kopple KD. Molecular properties that influence the oral bioavailability of drug candidates. J Med Chem. 2002; 45:2615-2623.

Lipinski C, Hopkins A. Navigating chemical spaces for biology and medicine. Nature. 2004; 432:855-861.

Peterhans E, Schweizer M. BVDV a pestivirus inducing tolerance of the innate immune response. Biologicals. 2013; 41:39-51.

Klemens O, Dubrau D, Tautz N. Characterization of the determinants of NS2-3-independnet virion morphogenesis of pestiviruses. J Virol. 2015; 89:11668-11680.

Baginiski SG, Pevear DC, Seipel M, Sun SC, Benetators CA, Chunduru SK, Rice CM, Collett MS. Mechanism of action of a pestivirus antiviral compound. Proc Natl Acad Sci USA. 2000; 97:7981-7986.

Paeshuyse J, Leyseen P, Mabery E, Boddeker N, Vrancken R, Froeyen M, Ansari IH, Dutartre H, Rozenski J, Gil LH, Letellier C, Lanford R, Canard B, Koenen F, Kerkhofs P, Donis RO, Herdewijn P, Watson J, De Clercq E, Puerstinger G, Neyts JA. novel, highly of pestivirus replication that targets the viral RNA-dependent RNA polymerase. J virol. 2006; 80:149-160.

Musiu S, Leyssen P, Froeyen M, Chezal JM, Neyts J, Paeshuyse J. 3-(imidazo[1,2-a: 5,4-b']dipyridin-2-yl) aniline inhibits pestivirus replication by targeting a hot spot drug binding pocket in the RNA-dependent RNA polymerase. Antiviral Res. 2016; 129:99-103.

Puerstinger G, Paeshuyse J, De Clercq E, Neyts J. Antiviral 2,5-disubstituted imidazo[4,5-c]pyridine from anti-pestivirus to anti-hepatitis C virus activity. Bioorg Med Chem Lett. 2007; 17:390-393.

Puerstinger G, Paeshuyse J, Heinrich S, Mohr J, Schraffi N, De Clercq E, Neyts J. Antiviral 2,5-disubstituted imidazo[4,5-c]pyridine further optimization of anti-hepatitis C virus activity. Bioorg Med Chem Lett. 2007; 17:5111-5114.

Vrancken R, Paeshuyse J, Haegeman A, Puerstinger G, Froeyen M, Herdewijn P, Kerkhofs P, Neyts J, Koenen F. Imidazo[4,5-c]pyridines inhibit the in vitro replication of the classical swine fever virus and target the viral polymerase. Antiviral Res. 2008; 77:114-119.