Original Article
Carrier testing for autosomal recessive hereditary disorder in Korean proven bulls
Kyu-Sang Lim, Byung-Ho Park, Tae-Jeong Choi, Dajeong Lim*

, Yong-Min Cho*
National Institute of Animal Science, RDA, Wanju, 55365, Republic of Korea
*Corresponding author: Dajeong Lim, National Institute of Animal Science, RDA, Wanju, 55365, Republic of Korea +82-63-238-7306, +82-63-238-7347,
lim.dj@korea.kr Yong Min Cho, National Institute of Animal Science, RDA, Wanju, 55365, Republic of Korea +82-63-238-7301, +82-63-238-7347,
variance@korea.kr
© Research Institute of Veterinary Medicine, Chungbuk National University. All rights reserved.
Received: Nov 3, 2016; Revised: Nov 16, 2016; Accepted: Nov 30, 2016
Abstract
Carrier testing for autosomal recessive hereditary disorders in the elite sire population has great significance for the domestic animal breeding. Because the recessive allele embedded in carriers without clinical signs may be passed to the next generation and rapidly spread throughout the population. The occurrences of various autosomal recessive hereditary disorders have been reported, and several causative mutations were elucidated in cattle. However, there is no report for the hereditary disorders in Korean cattle (Hanwoo) although Hanwoo is the indigenous purebred in Korea and have been improved by the national breeding programs in the last 30 years. Here, we investigated the presence of carrier for the following hereditary disorders in the Korean proven bulls (n=78; 42 family) using DNA based analysis: Chediak–Higashi syndrome, spherocytosis, claudin-16 deficiency, factor XI deficiency. The causative genes for these diseases (lysosomal trafficking regulator, solute carrier family 4 member 1, Claudin-16 and coagulation factor XI, respectively) were analyzed by polymerase chain reaction and direct sequencing. As a results, there was no carrier individual, and all animals were normal. Although the recessive alleles for four disorders were not identified in this study, further investigation for other hereditary disorders still remains to remove deleterious factors in the genetic improvement of Korean cattle.
Keywords: Hanwoo; Chediak–Higashi syndrome; spherocytosis; claudin-16 deficiency; factor XI deficiency
Introduction
상염색체 열성유전질병(autosomal recessive hereditary disorder)이란 원인유전자(causative gene)가 상염색체 (autosome)에 위치하며, 열성대립유전자(recessive allele) 를 2개 가지고 있을 때 임상증상으로 나타나는 질병을 말한다[1]. 특정 원인유전자의 돌연변이에 의해 발생하며, 열성대립유전자를 통해 후대에 전달이 될 수 있기 때문에 single gene disorder 또는 Mendelian disorder 라고도 불 린다.
가축의 경우 유전능력 개량을 위해 강력한 선발압력(selection pressure)속에 근친교배(inbreeding)가 이루어진 다. 이러한 근친교배는 집단내 동형접합체(homozygote)의 빈도를 높이므로 유전질병에 대한 위험도를 증가시킬 수 있 다. 상염색체 열성유전질병의 경우 열성대립유전자가 동형 접합체가 되었을 때 임상증상이 발현되기 때문에 매우 드물 게 나타난다. 이형접합체(heterozygote)를 가지고 있는 개 체는 보인자(carrier)로서 표현형적으로는 정상개체와 구분 이 안되는 한계가 있었지만, 최근 분자유전학적인 기술의 발 달과 함께 DNA수준에서 개체별 유전자형을 확인할 수 있 게 되었다.
한국 고유 소품종인 한우(Hanwoo)의 경우, 지난 30여년간 후대검정(progeny test)를 통한 한우 보증씨수소(Korean proven bulls) 선발로 유전적 개량을 해왔다[2]. 그러나, 아 직까지 한우 집단내 유전질병에 대한 모니터링 체계가 갖추 어지지 않았기 때문에 유전질병관련 원인유전자 혹은 원인돌 연변이에 대한 연구가 전무하다[3]. 특히, 보인자 개체가 있 을 경우 임상적으로 유전질병증상이 관찰되지 않더라도 집 단내 열성대립유전자의 빈도가 증가할 수 있기 때문에, 한우 보증씨수소 집단에 DNA분석을 통한 보인자 검사(carrier testing)는 매우 중요한 의미를 가진다.
아직까지 한우에서는 보고된 유전질병이 없다. 이에 단일 품 종으로서 마블링 중심으로 개량 되어온 일본흑우(Japanese black cattle)에서 보고된 4가지 주요 상염색체 열성유전질 병(Chediak-Higashi syndrome [4], spherocytosis [5], claudin-16 deficiency [6], factor XI Deficiency Syndrome [7])에 대해 보인자 검사를 실시했다.
Chediak–Higashi syndrome (CHS)은 lysosomal trafficking regulator (LYST) 유전자의 돌연변이(mutation)에 의해 발 생하는 유전질병으로[8], 사람과 쥐에서 잘 알려져 있을 뿐 만 아니라[9], 소[10] 에서도 보고되었다. 소의 LYST 유전 자는 염색체 28번(BTA 28)에 위치하고 있으며, exon 19 영역내 단일염기다형성(single nucleotide polymorphism, SNP)으로 인해 아미노산 서열이 히스티딘(histidine, 정상 개체)에서 아르기닌(arginine, CHS발병개체)으로 변화함 으로써 CHS를 유발한다. 소에서 Hereford [11], Brangus [12], Japanese black cattle [4] 등 다양한 품종에서 CHS 가 보고되었으며, 임상증상의 정도가 품종별로 차이가 있다 고 보고되었다. 발병개체는 부분백색증(partial albinism)으 로 인해 옅은 모색을 나타내며, 면역력감소로 인해 반복적인 박테리아 감염과 함께, 출혈(bleeding tendencies) 증상을 수반하는 특징이 있다[13].
Spherocytosis(erythrocyte membrane protein band III deficiency)는 band 3라고 하는 적혈구단백질을 코딩 하고 있는 solute carrier family 4 (anion exchanger), member 1 (SLC4A1) 유전자에 돌연변이가 생겨 발생한다[14]. SLC4A1 유전자는 band 3 또는 anion exchanger 1 (AE1) 라고 도 불린다. 사람의 AE1 유전자에서는 missense, nonsense, frameshift 돌연변이 등 다양한 변이가 알려져 있으며, 소에 서는 exon 17번에 위치한 nonsense 돌연변이가 동형접합 체가 될 때 발병하여 열성유전양식을 나타낸다[5]. 이 질병 의 경우 선천적인 용혈성빈혈(congenital hemolytic anemia) 증상과 함께 성장지연 증상을 나타낸다[5, 14].
Claudin-16 deficiency는 claudin-16 (CLDN16) 유전자의 결실돌연변이(deletion mutation)에 의해 발병한다[6, 15]. Claudin-16 단백질은 세포결합형태 중 밀착연접(tight junction)의 주요 구성인자로서 세포간 간극을 밀착시키고 세포극성을 유지하는데 중요한 역할을 하는데, 결실돌연변 이는 exon 1~4번 전체와 exon 5번의 일부분을 포함하고 있 어 정상적으로 단백질을 만들지 못하게 한다. Claudin-16 deficiency가 발병하면 세뇨관형성장애(renal tubular dys plasia)를 가져와 만성 신부전증(chronic renal failure) 및 만성 간질성신염(chromic interstitial nephritis)을 유발하 게 된다[16].
제XI인자결핍증(factor XI deficiency)은 coagulation factor XI (F11) 유전자의 exon 12번에 76 bp 크기의 서열이 삽 입(insertion)되는 돌연변이로 인해 stop codon이 중간에 생김으로써 발병한다[17]. Factor XI (plasma thromboplastin antecedent) 단백질은 혈액응고에 관여하는 주요 단 백질 중에 하나이기 때문에, 제XI인자결핍증이 발병한 개체 는 출혈 및 빈혈증상을 보이며, 사람, 개, 소 등에서 보고되 었다[18]. 송아지의 경우, 태줄에서 출혈이 나기도 하며, 단 각(dehorning), 거세(castration)시 삼출성 출혈이 지속되 는 경우가 있고, 암소에서는 핑크색 초유가 나타나기도 한다.
본 연구에서는 한우 보증씨수소 78두(총 42 가계)에 대 해 위에서 기술한 상염색체 열성유전질병관련 보인자 검사를 실시하여, 한우 집단내 유전질병관련 돌연변이(causative mutation)가 존재하는지 검증했다.
Materials and Methods
공시축
본 연구에서 사용된 공시축은 농협중앙회 한우개량사업소, 도 축산연구센터, 한우육종농가, 국립축산과학원에서 능력 검정을 실시한 집단에서 당대 41~62차, 후대 44~56차에 선발된 한우 보증씨수소 78두이다. 한우 보증씨수소 78두는 2003년부터 2011년에 출생한 개체로써, 혈통 분석 결과 한 우 보증씨수소 집단의 42개 가계에 분포되어 있었다. 한우보 증씨수소 78두의 정액으로부터 genomic DNA를 추출하여 실험을 수행하였다. 동물실험은 축산과학원 동물실험윤리위 원회(Institutional Animal Care and Use Committee)의 승인(승인번호 NIAS2015-774)하에 수행되었다.
유전자형 분석용 primer 제작
각 질병에 대한 개체별 유전자형분석에는 polymerase chain reaction (PCR) 및 direct sequencing을 이용하였 으며, 이를 위해 특이 primer 세트를 제작했다.
소의 LYST 유전자(accession number: AC_000185.1) 는 BTA 28에 위치하고 있으며, 총 58개의 Exon으로 구 성되어 있다. Exon 19에 위치한 SNP (c.6044A>G)에 대 해 direct sequencing을 이용한 유전자형분석을 위해 특이 primer 세트(P1)를 제작했다(Table 1).
Table 1.
Primer sets for PCR amplification
|
Target gene |
Primer set |
Primer sequence (5'→3') |
Annealing temperature (°C) |
Size (bp) |
|
LYST
|
P1 |
Forward: |
GGGGAGGTGGAGTTGGAATA |
60 |
886 |
|
|
|
Reverse: |
GCACAGAGGCCATTCAATGA |
|
|
|
AE1
|
P2 |
Forward: |
AGTAAGTTCCCAAGTGCCCA |
58 |
660 |
|
|
|
Reverse: |
GTTTGTCCTTCGTTGCCCTT |
|
|
|
CLDN16
|
P3 |
Forward: |
TATGCTGTTGATGTTTATGTAG |
57 |
375 |
|
|
|
Reverse: |
CCCCCCCCCGCCTTTTTC |
|
|
|
|
P4 |
Forward: |
ATTGTATTTTTAGGAGTGACTC |
57 |
722 |
|
|
|
Reverse: |
CCCCCCCCCACTCTATAC |
|
|
|
|
P5 |
Forward: |
AATGCACTCATTTCTCCAGTTCAGC |
57 |
800-900 |
|
|
|
Reverse: |
GTCGACAGCATACATTTTGGCAGTC |
|
|
|
F11
|
P6 |
Forward: |
CCCACTGGCTAGGAATCGTT |
52 |
244 or 320 |
|
|
|
Reverse: |
CAAGGCAATGTCATATCCAC |
|
|
Download Excel Table
소의 AE1 유전자(accession number: AC_000176.1)는 BTA 19에 위치하고 있으며, 총 21개의 Exon으로 구성되 어 있다. Exon 17에 위치한 SNP (c.1990C>T)에 대한 direct sequencing을 위해 특이 primer 세트(P2)를 제작 했다.
소의 CLDN16 유전자(accession number: AC_000158.1) 는 BTA 1에 위치하며 5개의 Exon으로 구성되어 있다. CLDN16 은 InDel(insertion / deletion, 삽입결실) 양상에 따라 두 종류의 돌연변이형(mutant type)을 가진다. Type 1 mutant의 경우, Exon 1번 ~ Exon 4번을 포함하는 약 37 kb의 서열이 deletion 되고, 그 위치에 31 bp 서열이 insertion 된다. Type 2 mutant의 경우에는, 더 많은 영역인 56 kb의 서열이 deletion 되며 이 영역은 Exon 1 ~ Exon 4번 뿐만 아니라 Exon 5번의 앞부분 21 bp를 포함하고 있다. 따 라서, 본 연구에서는 wild type과 두 mutant type을 구분하 기 위해 기존에 보고되어 있는 3종류의 primer 세트(P3 ~ P5)를 이용하였다[6, 15].
소의 F11 유전자(accession number: AC_000184.1) 는 BTA27에 위치하며 15개의 Exon으로 구성되어 있다. Factor XI deficiency 발병개체는 정상개체와 비교하여 Exon 12번에 76 bp 길이의 서열이 insertion되어 있으며, 이로 인해 stop codon이 중간에 생겨 정상적인 F11 단백질 을 만들 수 없다. 유전자형 분석을 위해 Marron 등이 보고한 primer 세트(P6)를 이용했다[17].
PCR 및 direct sequencing을 통한 유전자형분석
PCR은 총 부피 20 μL로, genomic DNA (30 ng/μL) 1 μL, forward/reverse primer (10 pmol/μL) 각각 1 μL씩, 2.5 mM dNTP 2 μL, 10X PCR buffer 2 μL, Taq polymerase 0.2 μL를 혼합했다. PCR 반응조건은 95℃에서 12분 predenaturation 실시한 다음, 95℃에서 30초, Table 1에 primer 세트별로 제시된 annealing 온도에서 40초, 72℃ 에서 30초씩 총 33회 증폭시키고, 끝으로 72℃에서 7분간 last-extension을 실시했다. PCR 종료후 1%의 agarose gel 에서 150 V로 30분간 전기영동(electrophoresis)을 실시하여 증폭여부 및 크기를 확인했으며, 증폭된 PCR산물 의 크기는 각각 Table 1에 나타나 있다.
InDel형태로 확인되는 claudin-16 deficiency 및 factor XI deficiency의 경우 PCR을 통해 유전자형을 확인할 수 있 었으며, CHS와 구상적혈구증의 경우 SNP 유전자형확인을 위해 direct sequencing을 실시했다.
증폭된 PCR산물에 대한 Sequencing에는 Big Dye Cycle Sequencing Ready Reaction Kit V3.0 (Life Technologies, Carlsbad, CA, USA)와 ABI PRISMⓇ 3730 Genetic Analyzer (Life Technologies)를 이용했다. Sequencing 결과에 대해 SeqMan (DNASTAR, Madison, WI, USA)를 이용하여 alignment한 후 각 개체별 유전자형을 분석했다.
Results
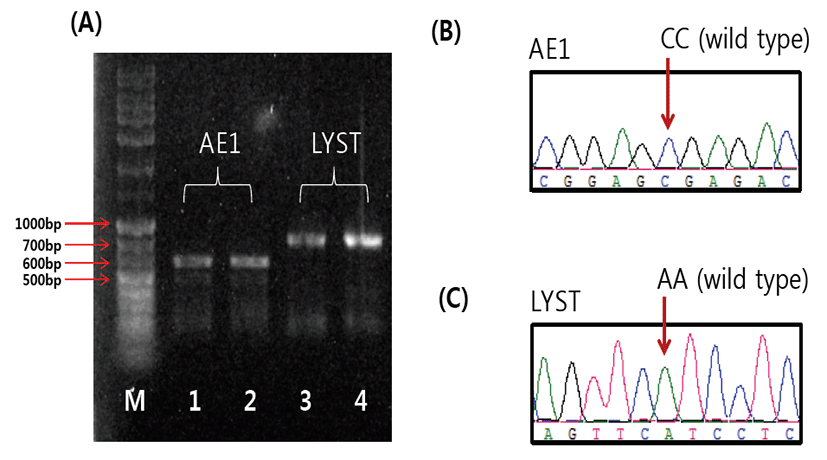
CHS의 경우, 프라이머 세트 P1을 이용하여 LYST 유전자 의 SNP (c.6044A>G)를 포함하는 886 bp의 PCR산물을 얻을 수 있었다(Fig. 1). 이 SNP의 대립유전자 G 는 mutant type으로, 이것을 두 개 가지고 있는 동형접합체 개체 는 CHS가 발병한다. 본 연구에서, 각 개체별 PCR 산물에 대한 direct sequencing 결과 모두 AA 유전자형을 가지 고 있는 것으로 나타났다(Table 2). 따라서, 본 연구에 이 용된 보증씨수소 집단에는 보인자(carrier) 개체가 없는 것 을 확인했다.
Fig. 1.
The electrophoresis image (A) and sequence chromatograms of AE1 (B) and LYST (C). The sizes of PCR products for AE1 and LYST genes are 660 bp (line 1~2) and 886 bp (line 3~4), respectively. The arrows in the electrophoresis image show the size of DNA ladder (1kb+) fragment.
Download Original Figure
Table 2.
Genotype and allele frequencies for the variations related
|
Disorder |
Gene |
N |
Genotype |
Allele |
|
Chediak–Higashi Syndrome |
LYST
|
78 |
WW1 |
WM |
MM |
W |
M |
|
|
1.00 |
0.00 |
0.00 |
1.00 |
0.00 |
|
|
(78)2 |
(0) |
(0) |
|
|
|
|
Spherocytosis |
AE1
|
78 |
WW |
WM |
MM |
W |
M |
|
|
1.00 |
0.00 |
0.00 |
1.00 |
0.00 |
|
|
(78) |
(0) |
(0) |
|
|
|
|
Claudin-16 deficiency |
CLDN16
|
78 |
WW |
M1M1 |
M2M2 |
W |
M1 |
M2 |
|
1.00 |
0.00 |
0.00 |
1.00 |
0.00 |
0.00 |
|
(78) |
(0) |
(0) |
|
|
|
|
Factor XI Deficiency Syndrome |
F11
|
78 |
WW |
WM |
MM |
W |
M |
|
|
1.00 |
0.00 |
0.00 |
1.00 |
0.00 |
|
|
(78) |
(0) |
(0) |
|
|
|
Download Excel Table
Spherocytosis와 연관되어 있는 AE1 유전자내 SNP (c.1990C>T)에 대한 유전자형 분석결과, 유사한 결과를 확인할 수 있었다. 분석에 이용된 전 개체들은 CC 유전자형 으로 wild type 대립유전자를 2개 가지고 있는 동형접합체 로 나타났다(Table 2).
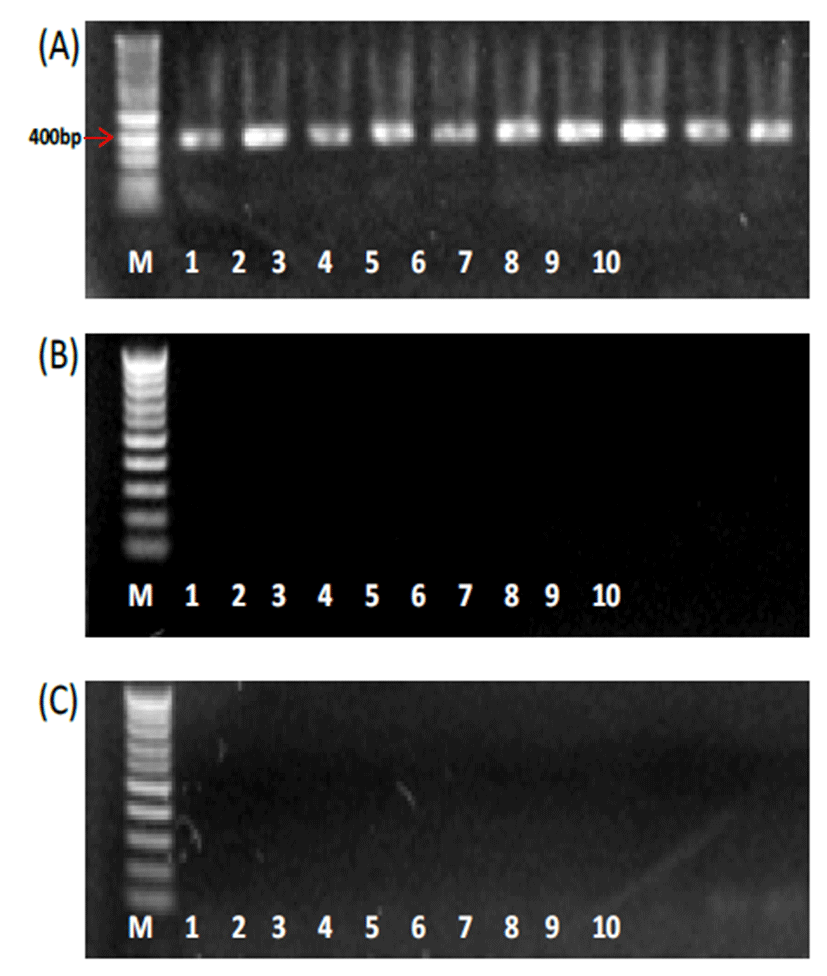
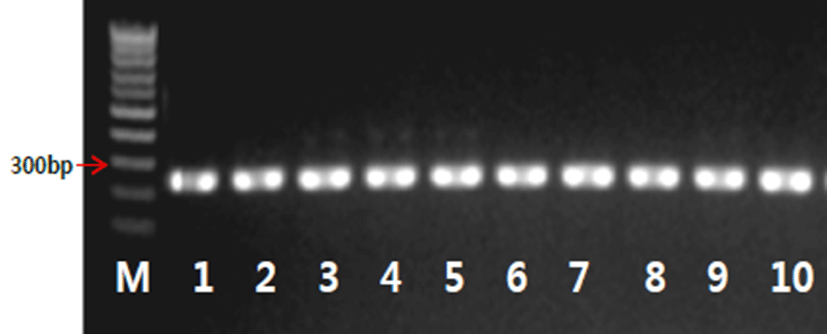
각각 claudin-16 deficiency 와 factor XI deficiency syndrome에 연관되어 있는 CLDN16 및 F11 유전자들 의 InDel 패턴을 분석한 결과, 전 개체에서 모두 wild type allele을 2개 가지고 있는 동형접합체로 나타났다(Table 2). CLDN16의 경우에는 wild type과 함께 두 종류의 mutant type을 구분하기 위해 각각 특이적인 프라이머 세트 (P3~P5)를 이용하였는데, wild type allele을 탐지할 수 있 는 P3에서만 PCR밴드를 확인할 수 있었다(Fig. 2). PCR 이 증폭되지 않은 프라이머 세트 P4 및 P5의 경우 추후에 positive control을 대상으로 PCR test를 실시하는 것이 필 요할 것으로 판단된다. 이런 한계가 있음에도 불구하고, 본 연구에 이용된 CLDN16 프라이머 세트는 일본흑우에서 발 병개체, 보인자개체, 정상개체를 대상으로 검증된 프라이머 세트이고[6, 15], 이중 P3은 실제 PCR증폭이 되었기 때문 에, P4 및 P5가 증폭되지 않은 이유는 PCR조건의 문제가 아 니라 positive animal이 집단내 존재하기 않기 때문으로 판 단할 수 있다. 따라서, 본 한우 보증씨수소 집단은 모두 wild type으로 나타났다. F11 에서 PCR 산물의 크기가 wild type 의 경우 244 bp, mutant type에서 320 bp으로 나타나는데, 한우 보증씨수소 집단에서는 모두 244 bp의 PCR밴드만 확 인되었다(Fig. 3).
Fig. 2.
The electrophoresis images for PCR products of CLDN16 gene. (A) PCR with 375 bp for the wild type using primer set P3. (B) PCR for the type 1 mutant using primer set P4 (expected size: 722 bp). (C) PCR for the type 1 mutant using primer set P5 (expected size: 800 to 900 bp). PCR products for primer set P4 and P5 were not amplified in KPN population used in this study. M, 1kb+ DNA size marker; line 1~10, individuals.
Download Original Figure
Fig. 3.
The electrophoresis images for PCR product for F11 gene. Expected sizes of PCR products for wild and mutant types were 244 bp and 320 bp, respectively. M, 1kb+ DNA size marker; line 1~10, individuals.
Download Original Figure
정 등[3]이 한우 22두에 대해 PCR-restriction fragment length polymorphism (PCR-RFLP)방법을 이용하 여 CHS와 spherocytosis관련 유전자형분석을 실시했으며, 이 연구에서도 본 연구의 결과와 동일하게 모두 정상(wild type)으로 나타났다.
일본흑우의 사례에서는 CHS가 보고된 초기에는 매우 높 은 빈도(Ogawa 등[4], 28%; Kunieda 등[10], 45%; Yamakuchi 등[19], 40%)로 발병했으며, 최근 연구[20]에 서는 보인자개체(carrier, heterozygote)만 9%가 나타났 다. 이는 지속적인 CHS열성대립유전자를 제거하는 방향으 로 선발되었기 때문으로 생각되며, 그럼에도 불구하고 집단 내에 임상증상이 없는 보인자형태로 존재할 수 있음을 알 수 있다.
Ohba 등[21]이 임상증상이 없는 일본흑우집단에 대해 F11 유전자에 대한 유전자형 분석을 실시하여, mutant allele의 동형접합체 개체가 6%, 이형접합체 개체가 41%로 나타나 집단내 mutant allele의 빈도는 26%로 나타났다고 보고하면서, factor XI deficiency syndrome이 일본흑우 에 널리 퍼져있을 가능성을 언급했다. Patel 등[22] 은 다양 한 품종(Hereford 및 zebu, water buffalo)에 대해 분석을 실시했으며, Hereford에서만 2마리(0.6%)의 보인자개체 로 확인되었고 다른 품종에서는 모두 정상개체로 나타났다.
Discussion
현재까지 사람에서는 5000개가 넘는 상염색체 열성유전질 병들이 알려져 있으며, 소에서는 이보다 적지만 약 200여개 가 알려져 있다[23]. 유전질병관련 데이터베이스인 Online Mendelian Inheritance in Animals (OMIA)에서는 현재까 지 소에서 원인유전자가 밝혀진 형질 또는 유전질병(118개, 2016년 7월 기준)을 제공하고 있다. 전세계적으로 보편적 으로 이용하고 있는 인공수정(artificial insemination)방법 은 유전적 개량을 극대화 할 수 있는 장점과 함께 정상적인 표현형을 가진 보인자개체로 인한 유전질병이 확산될 수 있 는 위험성도 같이 내재하고 있다[24]. 따라서, 선발된 씨수 소에 대한 보인자검사는 매우 중요한 의미를 가진다.
소에서 유전질병들은 품종특이적으로 나타나는 것으로 보 고되어 있다. 일본흑우는 본 연구에서 분석한 4가지 질병 외 에도 chondrodysplastic dwarfism, hemophilia A 등이 보 고 되어 있으며[25], Holstein에서는 complex vertebral malformation, bovine leukocyte adhesion deficiency syndrome 등이 보고되었다[26]. 아직까지 한우에서는 유 전질병발병관련 보고가 없었다. 따라서, 향후 한우에서도 선 발된 보증씨수소들에 대한 유전질병관련 형질자료 수집 뿐 만 아니라 보인자를 선별할 수 있는 체계마련이 필요할 것 으로 판단된다.
한우는 매년 20여두의 보증씨수소를 선발하여 전국 농가 에 보급하는 체계를 가지고 있기 때문에, 보증씨수소의 유전 질병여부는 한우개량에 있어 매우 중요한 의미를 가진다. 본 연구에서는 원인유전자가 알려져 있는 Chediak-Higashi syndrome, spherocytosis, claudin-16 deficiency, factor XI deficiency syndrome에 대해 DNA기반 유전자형분 석을 실시하여 그동안 선발된 한우보증씨수소 집단내에 유 전질병 요인이 존재하는지 검증했다. 그 결과, 분석에 이용 된 78두 전개체에서 모두 정상(wild type)으로 나타났다. 본 연구에서 분석한 유전질병은 일본흑우에서 보고된 것으 로, 유전질병패턴이 한우집단과 상이할 수 있으므로 이 4개 질병뿐만 아니라 다른 유전질병에 대한 모니터링에도 지속 적인 관심이 필요하다.
Acknowledgements
본 연구는 농촌진흥청 연구사업(PJ01134902)및 국립축 산과학원 박사후연수과정 지원사업으로부터 연구비를 지원 받아 수행하였으며, 연구비 지원에 감사 드립니다.
References
Patel R. Autosomal recessive genetic disorders of cattle breeds Worldwide-a Review. Journal of Livestock Biodiversity. 2010; 2:35-41.

Kim N.S., Lee J.J., Dang C.K., Jung Y.H., Cheon H.J. Prospect and retrospect on improvement of Hanwoo (Korean cattle) for 50 Years. Bulletin of the Animal Biotechnology. 2014; 6:1-13.

Chung H.J., Yu S.L., Sang B.C., Lee J.H. A Fundamental Genetic Study for Identifying Band 3 and CHS Genetic Diseases in Korean Cattle (Hanwoo). Kor J Agri Sci. 2005; 32:53-61.

Ogawa H., Tu C., Kagamizono H., Soki K., Inoue Y., Akatsuka H., Nagata S., Wada T., Ikeya M., Makimura S. Clinical, morphologic, and biochemical characteristics of Chediak-Higashi syndrome in fifty-six Japanese black cattle. Am. J. Vet. Res. 1997; 58:1221-1226.

Inaba M., Yawata A., Koshino I., Sato K., Takeuchi M., Takakuwa Y., Manno S., Yawata Y., Kanzaki A., Sakai J. Defective anion transport and marked spherocytosis with membrane instability caused by hereditary total deficiency of red cell band 3 in cattle due to a nonsense mutation. J. Clin. Invest. 1996; 97:1804-1817.

Hirano T., Kobayashi N., Itoh T., Takasuga A., Nakamaru T., Hirotsune S., Sugimoto Y. Null mutation of PCLN-1/Claudin-16 results in bovine chronic interstitial nephritis. Genome Res. 2000; 10:659-663.

Kunieda M., Tsuji T., Abbasi A.R., Khalaj M., Ikeda M., Miyadera K., Ogawa H., Kunieda T. An insertion mutation of the bovine F11 gene is responsible for factor XI deficiency in Japanese black cattle. Mamm. Genome. 2005; 16:383-389.

Barbosa M.D., Nguyen Q.A., Tchernev V.T., Ashley J.A., Detter J.C., Blaydes S.M., Brandt S.J., Chotai D., Hodgman C., Solari R.C. Identification of the homologous beige and Chediak–Higashi syndrome genes. Nature. 1996; 382:262-625.

Barbosa M.D., Barrat F.J., Tchernev V.T., Nguyen Q.A., Mishra V.S., Colman S.D., Pastural E., Dufourcq-Lagelouse R., Fischer A., Holcombe R.F. Identification of mutations in two major mRNA isoforms of the Chediak- Higashi syndrome gene in human and mouse. Hum. Mol. Genet. 1997; 6:1091-1098.

Kunieda T., Nakagiri M., Takami M., Ide H., Ogawa H. Cloning of bovine LYST gene and identification of a missense mutation associated with Chediak-Higashi syndrome of cattle. Mamm. Genome. 1999; 10:1146-1149.

Burns G., Meyers K., Prieur D. Secondary amyloidosis in a bull with Chediak-Higashi syndrome. Can. J. Comp. Med. 1984; 48:113-114.

Ayers J., Leipold H., Padgett G. Lesions in Brangus cattle with Chediak-Higashi syndrome. Vet. Pathol. 1988; 25:432-436.

Introne W., Boissy R.E., Gahl W.A. Clinical, molecular, and cell biological aspects of Chediak–Higashi syndrome. Mol. Genet. Metab. 1999; 68:283-303.

Ito D., Koshino I., Arashiki N., Adachi H., Tomihari M., Tamahara S., Kurogi K., Amano T., Ono K., Inaba M. Ubiquitylation-independent ER-associated degradation of an AE1 mutant associated with dominant hereditary spherocytosis in cattle. J. Cell Sci. 2006; 119:3602-3612.

Hirano T., Hirotsune S., Sasaki S., Kikuchi T., Sugimoto Y. A new deletion mutation in bovine Claudin-16 (CL-16) deficiency and diagnosis. Anim. Genet. 2002; 33:118-122.

Hirayama H., Kageyama S., Moriyasu S., Hirano T., Sugimoto Y., Kobayashi N., Inaba M., Sawai K., Onoe S. Minamihashi. Genetic diagnosis of claudin-16 deficiency and sex determination in bovine preimplantation embryos. J. Reprod. Dev. 2004; 50:613-618.

Marron B., Robinson J., Gentry P., Beever J. Identification of a mutation associated with factor XI deficiency in Holstein cattle. Anim. Genet. 2004; 35:454-456.

Meydan H., Yildiz M.A., Özdil F., Gedik Y., Özbeyaz C. Identification of factor XI deficiency in Holstein cattle in Turkey. Acta Vet. Scand. 2009; 51:1-4.

Yamakuchi H., Agaba M., Hirano T., Hara K., Todoroki J., Mizoshita K., Kubota C., Tabara N., Sugimoto Y. Chediak-Higashi syndrome mutation and genetic testing in Japanese black cattle (Wagyu). Anim. Genet. 2000; 31:13-19.

Abdeen A., Sonoda H., Kobayashi I., Kitahara G., Ikeda M. A New Method for Rapid Detection of the Mutant Allele for Chediak-Higashi Syndrome in Japanese Black Cattle. J. Vet. Med. Sci. 2013; 75:1237-1239.

Ohba Y., Takasu M., Nishii N., Takeda E., Maeda S., Kunieda T., Kitagawa H. Pedigree analysis of factor XI deficiency in Japanese black cattle. J. Vet. Med. Sci. 2008; 70:297-299.

Patel R.K., Soni K.J., Chauhan J.B., Singh K.M., Sambasiva Rao K.R. Factor XI deficiency in Indian Bos taurus, Bos indicus, Bos taurus x Bos indicus crossbreds and Bubalus bubalis. Genet. Mol. Biol. 2007; 30:580-583.

Huston K. Heritability and diagnosis of congenital abnormalities in food animals. Vet. Clin. North Am. Food Anim. Pract. 1993; 9:1-9.

Patel R.K., Singh K.M., Soni K.J., Chauhan J.B., Rao K.R. Lack of carriers of citrullinaemia and DUMPS in Indian Holstein cattle. J. Appl. Genet. 2006; 47:239-242.

Kunieda T. Identification of genes responsible for hereditary diseases in Japanese beef cattle. Anim. Sci. J. 2005; 76:525-533.

Kumar V., Singh R.K., Sharma A. Deficiency of Uridine Monophosphate Synthase: A Recessive Disorder in Holstein Friesian Cattle. Vet World. 2010; 3:523-525.