모든 생물의 단백질합성에 필수적인 20종류의 아미노 아 실-tRNA-합성효소(aminoacyl-tRNA synthetase, ARS) 는 단백질합성 과정에서 특정 아미노산을 해당 tRNA에 공 유 결합시켜 아미노 아실-tRNA의 형성을 촉매 하는 효소이 다. 미생물과 같은 하등생물과 다르게 인간을 포함한 고등생 물에서는 아미노 아실-tRNA-합성효소가 세포 항상성 유 지를 위해 다양한 세포 신호 전달 네트워크에 관여하여 새 로운 기능을 수행하는 것으로 알려져 왔다 [1]. 새롭게 부가 된 세포 기능은 대부분 진화 과정 동안 생성된 효소기능과는 무관한 단백질 부위로부터 기인하는 것으로 보고되고 있다 [2]. 흥미롭게도 20종류의 효소 중에서 9종류의 합성 효소들 (aspartyl-, DRS; glutamyl-prolyl-, EPRS; isoleucyl-, IRS; lysyl-, KRS; leucyl-, LRS; methionyl-, MRS; glutaminyl-, QRS; arginyl-tRNA synthetase, RRS)은 효소 기능을 하는 단백질 도메인이 아닌 새롭게 확장된 단백질 도 메인 부위를 이용하여 3종류의 아미노 아실-tRNA-합성 효소 결합 다기능 단백질(aminoacyl-tRNA synthetaseinteracting multifunctional protein, AIMP1, 2, 3)과의 결 합을 통해 세포질 내에서 거대 복합체(multi-aminoacyltRNA synthetase complex, MSC)형태를 이루어 존재하고 있다 [3]. 현재 세포질내의 MSC 존재 이유는 명확하게 밝혀 지지 않았지만, 효율적인 단백질합성 효소로서의 기능과 더불 어 세포 항상성 조절에 중요한 역할을 한다는 사실이 밝혀지 고 있다 [4].
M S C 의 구성원으로 존재하는 합성효소들은 인산화 (phosphorylation)와 같은 전사후수식화(post-transla tional modification)과정을 거치면서 복합체로부터 방출되어 면역반응 [5], 암 성장 [6], 혈관신생 [7], 신진대사 [4] 등을 조 절하는 기능을 수행한다고 알려졌다.
MSC 구성원 중 EPRS는 유일하게 2종류의 합성효소 (glutamyl-tRNA synthetase; ERS, prolyl-tRNA synthetase; PRS)가 하나의 구조를 이루어 존재하는 효소 로, N-말단으로부터 글루타치온 S-전이효소(glutathione S-transferase, GST)와 유사한 형태를 가진 도메 인, ERS, WHEP[tryptophanyl-tRNA synthetase(W), histidyl-tRNA synthetase(H)와 EPRS(EP)에서 발견 되는 도메인으로 이로 인해 도메인 이름이 유래됨], 그리 고 PRS로 이루어진 1512개 아미노산으로 이루어진 효소단 백질이다 [8, 9]. 이전 연구에 의하면, EPRS는 인터페론- 감마 (interferon-γ, IFN-γ)가 처리된 세포에서 세린 886번과 999번이 인산화되고 MSC로부터 방출되어 [10], NS1-associated protein 1 (NSAP1), 60S ribosomal protein L13a (L13a), glyceraldehyde 3-phosphate dehydrogenase (GAPDH) 단백질들과 결합을 하여 인터 페론-감마-활성화된 번역 억제(IFN-γ-activated inhibitor of translation, GAIT)복합체를 형성한다고 보고되 었다 [5]. 이를 통해, GAIT 복합체는 염증반응 유전자들의 특정 mRNA 3’-untranslated region(UTR)의 2차 구조 를 인식하여 결합하고 리보솜과 mRNA 결합을 억제함으로 써 결과적으로 과도한 염증반응을 제어하는 기능을 한다고 밝혀졌다 [11-13].
최근 본 연구진은 EPRS가 항 바이러스 선천성 면역 (innate immunity)활성 인자로 작용한다는 새로운 기능을 규명 하였다 [14]. 바이러스에 감염된 세포내의 EPRS는 인터페 론-감마에 의한 인산화와는 다르게 세린 990번에서 특이적 인 인산화가 발생하여 MSC로부터 방출되는 것을 확인하였 다. 또한 프로테옴 분석으로부터 MSC로부터 방출된 EPRS 는 poly(rC)-binding protein 2 (PCBP2)라는 단백질과 결합함을 확인하였다. PCBP2 단백질은 항 바이러스 선천 성 면역반응 조절에 핵심이 되는 mitochondrial antiviralsignaling protein (MAVS)의 유비퀴틴화를 유도하여 프로 테오좀 분해를 촉진시키는 기능을 하는 것으로 알려져 있다 [15]. 본 연구진은 바이러스 감염 시 EPRS는 PCBP2와 결 합을 통해 경쟁적으로 MAVS와 PCBP 간의 상호작용을 차 단하고 결과적으로 PCBP2가 매개하는 MSVS 분해의 저해 를 통해 세포 내 안정성을 유도함으로써 항 바이러스 싸이토 카인 분비를 촉진함을 규명하였다 [14].
본 연구에서는, EPRS의 항 바이러스 면역 활성 기능을 연 구하는 과정에서 확보한 인플루엔자 A 바이러스가 감염된 인 간유래 기관지 상피세포(primary normal human bronchial epithelial cells)의 전사체 데이터를 활용하여, 바이러스 감 염 또는 인터페론-감마와 같은 외부 자극에 따라 특이적으 로 반응하여 나타나는 EPRS의 전사후수식화-의존적인 케 모카인 관련 유전자들의 발현 양상을 상세하게 분석, 비교함 으로써 이를 통해 외부 자극-특이적인 EPRS의 면역 반응 기능을 한층 더 확립하였다.
인간유래 기관지 상피세포[human bronchial epithelial 6 (HBE135-E6E7) cells (ScienCell Research Laboratories, USA)]는 keratinocyte serum-free medium에 5 ng/ ml human recombinant epidermal growth factor (EGF), 0.05 mg/ml bovine pituitary extract (BPE), 0.0058 mg/ml 인슐린, 500 ng/ml 하이드로코르티손 (hydrocortisone) 을 첨가하여 배양하였다. RAW264.7 (American Type Culture Collection, USA) 세포는 10% FBS, 100 unit/ml의 streptomycin과 penicillin이 첨가된 Dulbecco's Modified Eagle's Medium (DMEM) 배지를 사용하여 37℃, 5% CO2 조건에서 배양하였다. mRNA의 추출, 라이 브러리 제작 및 염기서열 분석을 위해 인간유래 기관지 상피 세포 (primary normal human bronchial epithelial cells) 로 분화시킨 후 인플루엔자 바이러스 (A/PR/8/34 influenza virus)를 1 MOI로 감염시켜 8시간과 24시간 배양하였다. 이 후 세포를 PBS 완충용액으로 모아 3000 rpm에서 3분간 원 심 분리하여 세포만 -80℃에서 보관하여 다음 실험에서 사 용하였다. 전체 RNA는 RNeasy RNA extraction Mini-Kit (Qiagen, Germany)로 분리하였고 아가로스 겔 전기영동으 로 RNA 품질을 확인하였다. TruSeq RNA sample preparation kit v2 (Illumina, USA)를 이용하여 poly-T oligo magnetic beads로 전체 RNA에서 mRNA를 정제하고 절편 화하여 cDNA로 전환하였다. Adapter에 cDNA를 연결한 후 중합연쇄반응을 통해 증폭시켰다. Hiseq-2000 (Illumina, USA)를 이용하여 Paired-end sequencing (101 × 2)을 수행하였고 각 과정은 두 번 반복 실험하였다. Homo sapiens 의 참조유전체 자료는 University of California Santa Cruz Genome Browser Gateway (assembly ID: hg19)로 부 터 얻어내었다. 참조유전체 색인은 SAMtools (v. 0.1.19) 과 Bowtie2-build component of Bowtie2 (v. 2.1.0)를 이용하여 만들었다. 이후 Tophat2 (v. 2.0)를 이용하여 참 조유전체 지도를 작성하였다. Cufflinks (v. 2.2.1) 을 사용 하여 46,895 RefSeq (UCSC hg19) 의 각 유전자를 정규화 하였고 Mev (v. 4.9.0) 으로 Heat maps을 제작하였다. R (v. 3.1.0) 과 PYTHON (v. 2.7.6) 으로 통계적 분석과 그래 프 구성을 하였다. 전사체 염기배열분석 (RNA-Seq) 결과 는 NCBI's Gene Expression Omnibus (GEO) accession code GSE75699에 등록하였다 [14].
케모카인 어레이(chemokine array) 분석을 위해 RAW264.7 세포에 각각 0.5 MOI의 인플루엔자 바이러스 를 감염시켰고, 인터페론-감마는 마우스 IFN-γ (KOMABIOTECH, Korea)를 0.5 μ g/ml를 처리하여 24시간 후 배양 상층액 만을 모아서 실험에 사용하였다. 배양 상층액 은 Proteome ProfilerTM Array Mouse Chemokine Array Kit ARY020 (R&D Systems, USA)을 사용하여 분 석하였다. 같은 양의 배양 상층액을 biotinylate된 검출항체 (detection antibody)와 함께 섞고 어레이 멤브레인에 뿌 려주었다. 4℃에서 하룻밤 동안 방치한 후, 세척버퍼(wash buffer)로 멤브레인에 붙지 않은 물질을 씻어내고, HRP가 달린 Streptavidin을 처리하였다. 상온에서 2시간 동안 처 리한 후 chemical reagent 1과 2를 각각 1:1로 섞어 뿌려주 었다. Davinch Chemi U400으로 이미지 확보 후 Totallab Quant v12.2에서 어레이 분석을 이용하여 강도를 분석하였 다. Reference spot으로 정규화하여 각 케모카인 값을 비 교하였다.
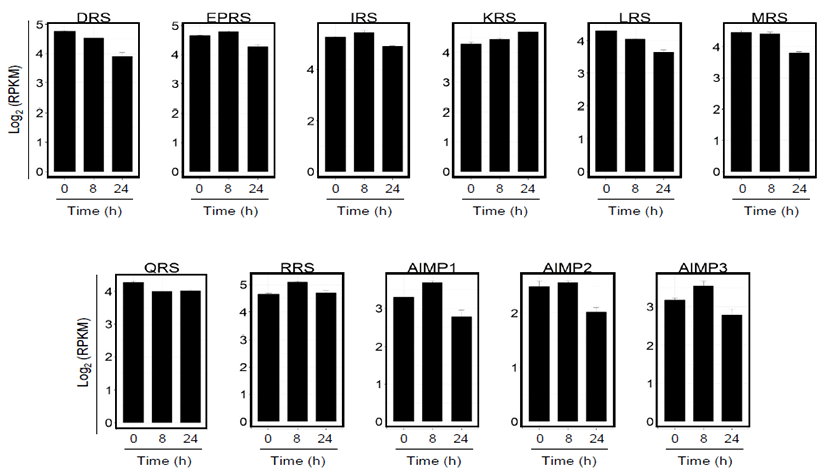
인간유래 기관지 상피세포에 인플루엔자 A 바이러스 (PR8)를 감염시켜 8시간과 24시간 후 세포 내 mRNA의 발 현양상을 전사체 염기배열분석 (RNA-Seq)을 통해 확인하 였다. 총 24,910개의 유전자 중 우선 MSC를 이루는 9종류 의 효소들과 AIMP1, 2, 3 mRNA 변화를 분석한 결과, 바이 러스 감염 후 시간이 경과함에 따라 유전자의 발현 양이 불 균등하게 변화되고 조절되는 양상을 확인할 수 있었다 (Fig. 1). KRS의 mRNA 발현은 바이러스 감염에 의해 꾸준히 증 가하는 반면에 DRS, LRS, MRS, QRS의 mRNA 발현은 다 소 감소하는 양상을 보였다. EPRS, IRS, RRS와 AIMP 단 백질들의 경우, mRNA는 바이러스 감염 8시간에서는 증가 하였으나 24시간에서는 감소함이 확인되었다 (Fig. 1). 이 는 MSC 구성 단백질들의 유전자들이 바이러스 감염에 반응 하여 세포 기능을 위해 발현이 조절되고 있음을 보여주는 결 과로 분석된다.
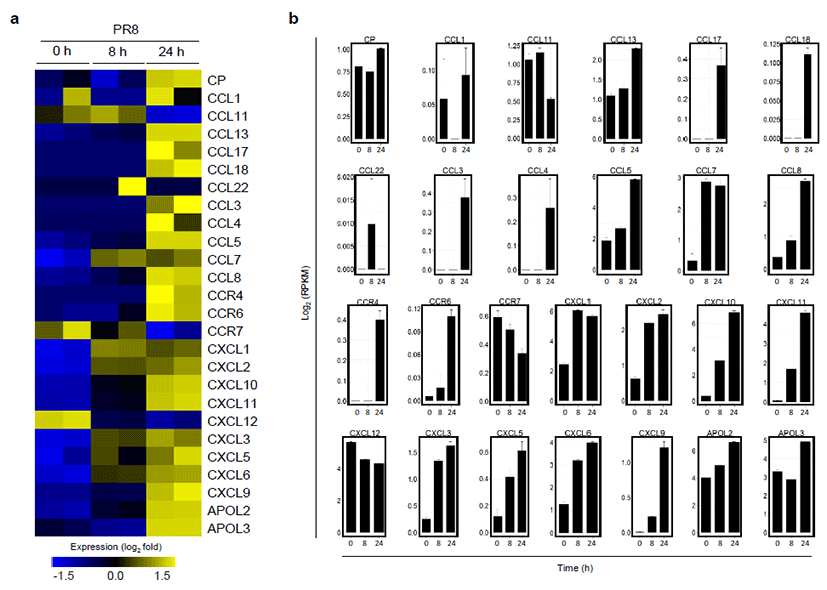
인터페론-감마 처리에 의해 EPRS가 GAIT 복합체 형성 시 발현이 억제된다고 알려진 케모카인과 케모카인 수용체 등의 발현 양상을 분석하였다. 그 결과 바이러스에 감염된 세 포의 케모카인과 관련된 유전자들의 발현은 전반적으로 증 가하는 것으로 나타났다 (Fig. 2). 특히 GAIT 복합체에 의 해 발현이 현저히 감소한다고 알려진 ceruloplasmin (Cp) 의 유전자도 [16] 바이러스 감염 시에는 오히려 증가함이 분석되었다. 이는 세포가 바이러스에 감염되면 EPRS가 인 터페론-감마 신호매개 세린 999번이 아닌, 새로운 아미노 산 잔기인 세린 990번이 인산화되고 이를 통해 항 바이러스 면역 반응 활성화를 유도한다는 결과와 일치하는 것으로 해 석된다 [14]. 즉, 바이러스 감염 특이적인 신호 전달에 의해 EPRS의 세린 990번이 인산화되면 GAIT 복합체 형성으로 인한 케모카인 관련 유전자의 발현 억제가 아닌, MAVS 중심 의 항 바이러스 싸이토카인과 케모카인 등의 발현을 오히려 증폭시킴을 나타낸다. 따라서, 본 연구를 통해 EPRS는 세포 에 전달되는 외부자극-특이적으로 케모카인 관련 유전자의 발현을 조절함을 확인하였다.
바이러스의 감염에 의한 mRNA 발현이 증가함에 따라 단 백질 발현 또한 증가하는지 확인하기 위하여 케모카인 어레 이 분석을 수행하였다. RAW264.7 세포를 인플루엔자 바이 러스 0.5 MOI로 감염시키거나 인터페론-감마 0.5 μg/ml 로 처리하여 24시간 후 확보한 세포 배양 상층액을 케모카 인 어레이에 반응시켰다. 그 결과 CCL6, IL-16, CXCL10, CCL2, CXCL2의 단백질 발현이 인터페론-감마 처리시 보 다 바이러스 감염에 의해서 상당히 증가함을 확인하였다 (Fig. 3). 이는 앞선 세포 내 mRNA의 발현 양상 분석과도 일치하는 결과로, 바이러스 감염 시, EPRS는 GAIT 복합체 에 의해 매개되는 신호 전달 경로와는 다른 바이러스 감염 특 이적인 신호전달 경로에서 선천성 면역 활성 인자로 역할을 수행함을 다시 한번 확인하는 결과이다.
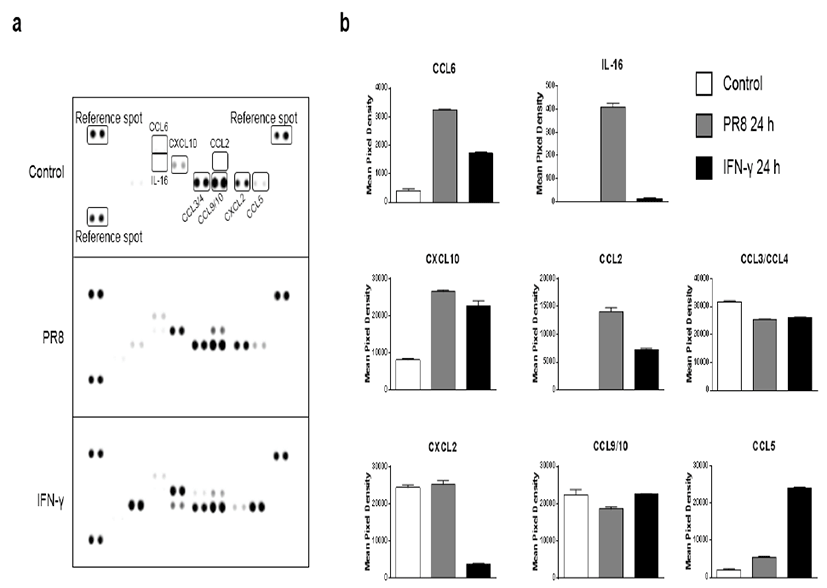
인터페론-감마에 의해 형성된 GAIT 복합체는 케모카인 mRNA와 리보솜간의 상호작용을 감소시킨다는 전사체 염 기서열 분석 결과가 보고된 바 있다 [17]. 이러한 결과를 본 연구에서 분석한 결과와 단순하게 비교하는 것은 무리가 있 을 수 있으나, 이전 논문에서 발현이 감소된다고 언급된 케 모카인 중 CCR6와 CCR4는 바이러스 감염 샘플에서는 증 가함을 확인할 수 있었다. 따라서 인터페론-감마 처리에 의 한 신호전달과 바이러스 감염 신호전달 체계에 따른 케모카 인 mRNA 발현 패턴의 차이가 있음을 알 수 있었다. 세포가 바이러스에 감염되면 즉각적으로 바이러스를 제거하기 위해 다양한 종류의 싸이토카인, 케모카인과 인터페론이 발현됨 이 잘 알려져 있다 [18-20]. 특히 1형 인터페론에 해당하 는 인터페론-α와–β는 병원체-연관성 분자패턴(pathogen- associated molecular pattern)을 인식하는 패턴인식 수용체(pattern recognition receptor)에 인지되어 분비되 고, 2형 인터페론에 해당하는 인터페론-감마는 적응면역계 (adaptive immunity)와 관련이 있다고 알려진 자연살해세 포(natural killer cell)등에 의해 분비되는 것으로 알려져 있 다 [21]. 따라서 EPRS는 바이러스 감염 초기에는 세린 990 번의 인산화를 통해 MSC로 방출되고 PCBP2와 결합하면서 MAVS를 안정화시켜 1형 인터페론과 케모카인 등의 발현을 증강시켜 선천성 면역 반응을 활성화 시키는 역할을 수행하 고, 이후 자연살해세포에 의해 인터페론-감마가 분비되면 세린 999번이 인산화되며 GAIT 복합체의 형성을 촉진하여 과도한 면역활성을 억제하기 위해 관련 유전자 mRNA들의 전사를 억제하는 기능을 수행하는 것으로 예측된다. 즉, 바이 러스를 제거하기 위해 분비된 항 바이러스성 물질이 체내에 서 지속적으로 발현되면 심각한 염증반응을 초래할 수 있기 때문에 EPRS는 GAIT 복합체 형성을 통한 전사조절을 통하 여 세포 내 항상성 유지에 관여 할 것으로 생각된다. 따라서, 감염된 인체내의 실제 상황에서는 바이러스 감염 정도와 시 간, 감염된 조직 부위 등에 따라 EPRS의 역할이 달라질 것 으로 보인다. 향후, 바이러스 감염 정도와 시간 등을 고려한 전반적인 EPRS의 기능 분석 연구가 수행되어야 할 것이다